Internationale Sammlung menschlicher Gene deckt jetzt 80 Prozent aller Bauanleitungen für Proteine ab – und steht Wissenschaftlern weltweit zur Verfügung
Die internationale ORFeome Collaboration (OC) stellt die weltweit größte öffentlich verfügbare Sammlung menschlicher Gene bereit. Alle wichtigen funktionellen Kategorien sind in der Sammlung vertreten. Mit jetzt 80 Prozent Abdeckung derjenigen Gene des Menschen, die die Bauanleitung für Proteine des Körpers enthalten, ist sie eine einzigartige und wertvolle Ressource für die Wissenschaft. Sie kann einen entscheidenden Beitrag zur Entdeckung von krankheitsrelevanten Veränderungen und von neuen Zielmolekülen für maßgeschneiderte Medikamente leisten.
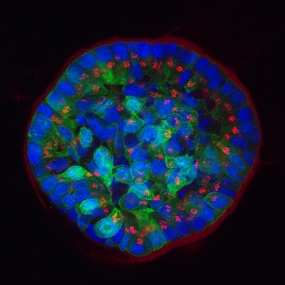
Baukasten menschlicher Proteine im Einsatz: Die Färbung von bestimmten Proteinen zeigt hier unkontrolliertes Wachstum von Zellen aus der Brustdrüse, eine Ursache für Krebserkrankungen der Brust. (Brustzellen in 3D Zellkultur gewachsen.)
© Aleksandra Balwierz, Abteilung Molekulare Genomanalyse, DKFZ
Obwohl nur etwa 1 Prozent der menschlichen Erbinformation Bauanleitungen für Proteine trägt, sind es diese, die das Schicksal von Zellen, Geweben und Organismen steuern. Wenn Wissenschaftler untersuchen, wie Proteine miteinander wirken oder beobachten, was passiert, wenn bestimmte Proteine in Modellorganismen vermindert sind oder ganz fehlen, lernen sie, Abläufe im Gesunden und Kranken zu verstehen. Solche Studien setzen eine möglichst vollständige Sammlung der Baupläne menschlicher Proteine voraus.
Die ORFeome Collaboration (OC), in der 13 internationale akademische und kommerzielle Arbeitsgruppen kooperieren, stellt eine solche Sammlung menschlicher Gene bereit. Diese umfasst jetzt 17.154 der insgesamt knapp 20.000 menschlichen Gene, die eine Bauanleitung für ein Protein enthalten. Professor Stefan Wiemann, Leiter der Abteilung Molekulare Genomanalyse am DKFZ in Heidelberg und Mitglied im Forschungs- und Förderprogramm e:Med sowie Mitglied des Nationalen Genomforschungsnetzes (NGFN, 2001-2013), das an der Finanzierung der OC beteiligt war, ist Erstautor der heute in der Zeitschrift Nature Methods erscheinenden Veröffentlichung. Der große Nutzen der OC Sammlung wurde bereits in einer Vielzahl von Studien gezeigt. Auch im e:Med Programm des Bundesforschungsministeriums (BMBF) wird die Ressource weit genutzt.
Die Sammlung enthält die einzelnen Bauanleitungen so bereit, dass die verschlüsselten Proteine einfach und präzise in einer großen Vielfalt von biologischen Systemen wie E. coli, Hefe und Säugetierzellen hergestellt und untersucht werden können. Alle wichtigen funktionellen Kategorien humaner Gene (z. B. Proteine des Stoffwechsels, Rezeptoren oder Transkriptionsfaktoren, die Gene aktivieren) sind in der Sammlung vertreten. Die Basenabfolge oder Sequenz eines jeden Gens wurde komplett ermittelt, nur vollständig verifizierte Materialien wurden in die Sammlung aufgenommen. Das im Rahmen des NGFN geförderte Deutsche cDNA Konsortium, das von Stefan Wiemann geleitet wurde, brachte mehr als 3.000 Gene in die OC ein, und hat damit den größten Beitrag von Partnern außerhalb der USA geleistet. Die Abteilung um Wiemann führt zudem alle bioinformatischen Analysen durch, die den Nutzern zeigen, welche Klone sich am besten für ihre jeweiligen Fragestellungen eignen. Die Forschergruppe aktualisiert diese bioinformatischen Analysen regelmäßig und macht diese über die Datenbank des Projekts (http://www.orfeomecollaboration.org) öffentlich zugänglich. Dort werden alle Informationen zu den jeweils abgedeckten Genen bereitgestellt, Wissenschaftler weltweit haben uneingeschränkten Zugang dazu.
„Die hohe Abdeckung des menschlichen Erbguts, kombiniert mit den vielseitigen Einsatzmöglichkeiten sowie dem freien Zugang zu der Sammlung macht sie zu einer einzigartigen und wertvollen Ressource für die wissenschaftliche Gemeinschaft. Sie hat das Potenzial, einen großen Beitrag dazu zu leisten, zahlreiche Krankheitsverläufe aufzuklären und damit neue Therapiemöglichkeiten zu entwickeln“, so Stefan Wiemann.
Originaltitel der Publikation:
The ORFeome Collaboration: A genome-scale human ORF-clone resource. Wiemann et al. Nature Methods 13, 191–192 (2016) doi:10.1038/nmeth.3776 Online-Veröffentlichung: 25. Februar 2016
Das Deutsche Krebsforschungszentrum (DKFZ) ist mit mehr als 3.000 Mitarbeiterinnen und Mitarbeitern die größte biomedizinische Forschungseinrichtung in Deutschland. Wissenschaftlerinnen und Wissenschaftler erforschen im DKFZ, wie Krebs entsteht, erfassen Krebsrisikofaktoren und suchen nach neuen Strategien, die verhindern, dass Menschen an Krebs erkranken. Sie entwickeln neue Methoden, mit denen Tumoren präziser diagnostiziert und Krebspatienten erfolgreicher behandelt werden können. Beim Krebsinformationsdienst (KID) des DKFZ erhalten Betroffene, Interessierte und Fachkreise individuelle Antworten auf alle Fragen zum Thema Krebs.
Um vielversprechende Ansätze aus der Krebsforschung in die Klinik zu übertragen und so die Chancen von Patientinnen und Patienten zu verbessern, betreibt das DKFZ gemeinsam mit exzellenten Universitätskliniken und Forschungseinrichtungen in ganz Deutschland Translationszentren:
Das DKFZ wird zu 90 Prozent vom Bundesministerium für Bildung und Forschung und zu 10 Prozent vom Land Baden-Württemberg finanziert und ist Mitglied in der Helmholtz-Gemeinschaft Deutscher Forschungszentren.
