Mechanismen der Genomkontrolle
- Funktionelle und Strukturelle Genomforschung
- Nachwuchsgruppe

Dr. Angelika Feldmann
Gruppenleiter
Die Gruppe „Mechanismen der Genomkontrolle“ erforscht, wie Gene in Raum und Zeit reguliert werden und wie diese Mechanismen bei Krebs fehlgeleitet werden.
Bild: Image: Visualization of 3D genome structure as Hi-C (top), Capture-C (middle) and cartoon (bottom),
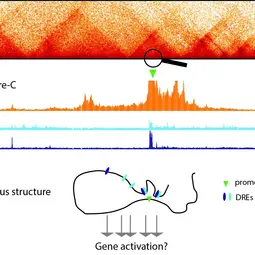
Bild: Image: Visualization of 3D genome structure as Hi-C (top), Capture-C (middle) and cartoon (bottom),
Unsere Forschung
Einer der faszinierendsten Prozesse in der Biologie ist die Entwicklung eines multizellulären Organismus mit über 200 verschiedenen Zelltypen aus einer einzigen Zelle. Das bedeutet, dass die meisten Zelltypen zwar das gleiche genetische Material haben, sich aber in Aussehen und Funktion unterscheiden. Solche Diversität kann nur durch exakte zeitliche und räumliche Genregulation erreicht werden. Hierfür müssen Genpromotoren Input von distalen genregulatorischen Elementen (distal gene regulatory elements, DREs), z. B. von Enhancern, integrieren. Trotz vieler Studien wissen wir immer noch wenig darüber, wie genau DREs Gene aktivieren. Wir nehmen an, dass diese oft hunderte von Kilobasen entfernt liegenden Elemente durch eine von einer Vielzahl an Proteinen regulierte Faltung der DNA (Schoenfelder et al., 2019, Feldmann et al., 2020, Rhodes et al., 2020) direkten Kontakt zu Genpromotoren aufbauen. Solche physischen Interaktionen korrelieren mit der Genaktivität und sind in Krebszellen oft verändert. Dies führte zu der Hypothese, dass sie für die Regulation von Genen in Entwicklung und Karzinogenese benötigt werden. Neuere Studien zeigen allerdings, dass physische Kontakte zwischen DREs und Genpromotoren auch aktivitätsunabhängig stattfinden können (Ghavi-Helm et al., 2014; Benabdallah et al., 2019, Feldmann et al., 2020), was nahelegt, dass sie Gene durch andere Mechanismen aktivieren.
Unser Ziel ist es die genauen Mechanismen zu verstehen, mit denen DREs während der Aktivierung und Aufrechterhaltung der Genaktivität mit Genpromotoren kommunizieren, mit einem besonderen Schwerpunkt auf der Bedeutung der physischen Kontakte in diesem Prozess. Wir kombinieren neueste genomische, proteomische und bioinformatische Methoden mit mittels Hochdurchsatztechniken produzierter genetischen Manipulationen, um genregulatorische Mechanismen in gesunden und pathologischen Entwicklungsprozessen zu erforschen.
PhD/Master/Postdoc-Bewerbungen nehmen wir per E-Mail entgegen. Bitte senden Sie uns ein Anschreiben und einen Lebenslauf mit 2-3 Referenzen.
Team
- Profil anzeigen

Dr. Angelika Feldmann
Gruppenleiter
- Profil anzeigen

Belinda Blum
Doktorandin
- Profil anzeigen

Sarah-Victoria Dachtler
Doktorandin
- Profil anzeigen

Dr. Ann-Kristin Dicke
Postdoktorandin
- Profil anzeigen

Gaia Oldfield
Master student
- Profil anzeigen

Sonja Prüssing
Technische Assistentin
- Profil anzeigen

Valeriia Smialkovska
Doktorandin
-
Ilayda Özcan
Ausgewählte Publikationen
S Mahara, S Prüssing, V Smialkovska, S Krall, S Holliman, B Blum, V Dachtler, H Borgers, E Sollier, C Plass and A Feldmann
Bitong Li, Yew Yan Wong, Neftali Flores-Rodriguez, Tara Davidson, Matthew S Graus, Valeriia Smialkovska, Hiroaki Ohishi, Angelika Feldmann, Hiroshi Ochiai, Mathias Francois
Maja C Funk, Jan G Gleixner, Florian Heigwer, Dominik Vonficht, Erica Valentini, Zeynep Aydin, Elena Tonin, Stefania Del Prete, Sylvia Mahara, Yannick Throm, Jenny Hetzer, Danijela Heide , Oliver Stegle, Duncan T Odom, Angelika Feldman, Simon Haas, Mathias Heikenwalder, Michael Boutros
Kontaktieren Sie uns

