Abteilung Funktionelle Genomanalyse
Dr. Jörg D. Hoheisel
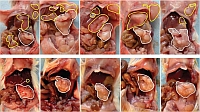
Potential des DRD2-Antagonisten Haloperidol – einem Molekül, das bereits zur Behandlung von Schizophrenie Verwendung findet – zur Behandlung von Pankreaskrebs. Repräsentative Bilder zeigen Tumoren in Mäusen, die nicht behandelt wurden (oben) oder nach einer Behandlung mit Haloperidol (unten). Weiße Linien verdeutlichen den Primärtumor, während gelbe Linien Metastasen aufzeigen. Eine Behandlung führte zu einem sehr starken Rückgang der Metastasenbildung.
© dkfz.de
Die Abteilung Funktionelle Genomanalyse untersucht, wie genetische Information in zelluläre Funktionen umgesetzt wird und wie diese Vorgänge reguliert werden.
Im Zentrum der Arbeiten stehen Untersuchungen an Bauchspeicheldrüsentumor-Material, Bauchspeicheldrüsen(Pankreas-)krebs gehört zu den tödlichsten Krebsformen überhaupt. Wir untersuchen Veränderungen der DNA-Sequenz, die epigenetische Modellierung des Genoms und das Bindungsverhalten von Transkriptionsfaktoren. Dies wird kombiniert mit Variationen der Transkriptmengen aller Gene, einschließlich nicht protein-kodierender RNA-Moleküle. Gleichzeitig analysieren wir die Unterschiede in der Proteinexpression. Diese Untersuchungen werden durch funktionelle Studien ergänzt, um tumor-relevante zelluläre Mechanismen zu identifizieren.
Neben dem grundlegenden Erkenntnisgewinn ist unser Ziel, Verfahren zur robusten, möglichst frühen und nicht-invasiven Diagnose zu etablieren, die auch für eine Krankheits-Prognose und Patientenstratifizierung sowie zur Überwachung des Behandlungserfolgs Anwendung finden können. Gleichzeitig suchen wir neue Therapieansätze.
Zurzeit liegt ein Fokus unserer Arbeiten auf dem Gebiet der Proteom-Analyse. Wir haben affinitätsbasierte Verfahren etabliert, deren Robustheit und Genauigkeit die Anforderung für eine klinische Anwendung erfüllen und in Tests überführt werden können, die den Vorgaben einer klinischen Routineanwendung genügen. Eine wissenschaftliche Fragestellung, die wir verfolgen, ist die Identifizierung krankheitsrelevanter Protein-Isoformen: strukturelle Veränderungen gehen oft mit veränderter Aktivität einher. Daneben untersuchen wir Protein-Interaktionen mit neuen Methoden, die ganz speziell auch die Untersuchung individueller Patienten erlauben. Ein dritter Schwerpunkt unserer Untersuchungen ist die Kartierung der protein-basierten Kommunikation zwischen den verschiedenen Zelltypen, die in einem Tumor und darüber hinaus vorliegen.
Seit einiger Zeit verfolgt die Abteilung neue biotechnologische Ansätze, die darauf abzielen, komplexe biologische Prozesse in vitro darzustellen. Eine Anwendung ist die Nutzung im Bereich der synthetischen Biologie zur Produktion von Bio-Molekülen. Eine zellfreie Biosynthese wird für viele biotechnologische und pharmakochemische Herausforderungen immer wichtiger. Ein zweites Ziel ist die Implementierung künstlicher molekularer Biosysteme. Sie ergänzen theoretisch-systembiologische Ansätze, um die Relevanz biologischer Modelle experimentell zu überprüfen. Ähnlich der Physik werden wir in Zukunft Erkenntnisse über zelluläre Funktionen in einem iterativen Prozess zwischen theoretischer und experimenteller Systembiologie gewinnen. Auf lange Sicht könnte dies zur Etablierung eines vollständig synthetischen, selbst-replizierenden Systems führen, mit der Perspektive, ein archetypisches Modell einer Zelle zu etablieren.
Die weiterführenden Seiten sind nur auf Englisch verfügbar.

