Molekulare Genomanalyse
- Funktionelle und Strukturelle Genomforschung

Prof. Dr. Stefan Wiemann
Head of Division
Krebs und andere Erkrankungen entstehen durch eine Vielzahl von Veränderungen in der Erbsubstanz, die entweder vererbt werden oder - insbesondere bei Krebs - in bis dahin normalen Körperzellen neu entstehen. Solche Veränderungen führen häufig zu Störungen in der Aktivität von Genprodukten, wodurch das Zusammenspiel in zellulären Signalwegen verändert wird, die das Zellwachstum eigentlich regulieren sollen. Diese Veränderungen möchten wir besser verstehen.
Bild: Brustzellen, die in einer 3D Matrix wachsen. Zellkerne (blau), Zytoskelet (grün) and Golgi (rot) sind gefärbt. © dkfz.de,
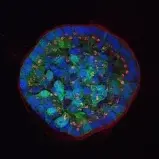
Bild: Brustzellen, die in einer 3D Matrix wachsen. Zellkerne (blau), Zytoskelet (grün) and Golgi (rot) sind gefärbt. © dkfz.de,
Unsere Forschung
Solche Prozesse können dann letztlich zu Krebs und den damit einhergehenden Krankheitsprozessen führen.
Ziel der Abteilung Molekulare Genomanalyse ist, die komplexen molekularen Mechanismen von Signalwegen mit Relevanz für die Krebsentstehung besser verstehen zu lernen und zu erkennen, wie Veränderungen auf die Tumorentstehung, Metastasen-Bildung und die Entwicklung von Resistenzen wirken. Um dies zu erreichen, führen wir Hochdurchsatz Analysen durch, in denen wir Zellsysteme mittels verschiedener bio-Werkzeuge gezielt beeinflussen und die dadurch hervorgerufenen Veränderungen messen. Dadurch konnten bereits Erkenntnisse über das Wirken von einzelnen Genprodukten in krebsrelevanten Signalwegen erhalten werden.
Auch unsere eigenen Arbeiten haben allerdings ergeben, dass im Verlauf der Krebsentwicklung meist mehrere Signalwege betroffen sind bzw. miteinander in Kontakt stehen. Dadurch können Krebszellen besser „lernen“ auf Therapeutika zu reagieren und deren erhoffte Wirkung zum Nachteil der Patienten zu umgehen. In Zukunft wollen wir daher Ansätze entwickeln, mit denen wir größere Netzwerke von Proteinen und anderen Faktoren untersuchen können. Weiterhin müssen Informationen, die im Reagenzglas generiert wurden, zunächst im Tiermodell und letztlich mithilfe von Patientenproben verifiziert werden, damit eine wirkliche Relevanz der aufgestellten Hypothesen nachgewiesen werden kann. Hier haben wir bereits einige Kooperationen aufgebaut, und die ersten erhaltenen Resultate sind viel versprechend. Diese Untersuchungen sollen uns erlauben, molekulare Marker für die Diagnose sowie mögliche Zielmoleküle für Therapien zu identifizieren.
Detaillierte Beschreibungen der wissenschaftlichen Projekte in der Abteilung sind auf der englischsprachigen Website verfügbar.
Team
- Profil anzeigen

Prof. Dr. Stefan Wiemann
Head of Division
-
Sarah Burmester
-
Daniela Fischer
-
Maike Hoesch
-
Dasaradha Jalapothu
-
Dr. Cindy Körner
-
Christos Nousias
-
Dr. Veronica Rodrigues de Melo Costa
-
Luisa Schwarzmüller
-
Rebeka Sharko
-
Carla Siebenkotten
-
Angelika Wörner
Ausgewählte Publikationen
Vlachawas E.-I. et al.
Sumer O. E. et al.
Radke J. et al.
Maia, A. et al.
Wahjudi, L.W. et al.
Full List of Publications
Kontaktieren Sie uns

