Bioinformatik der Genomik und Systemgenetik
- Funktionelle und Strukturelle Genomforschung

Prof. Dr. Oliver Stegle
Division Head
Unsere Forschungsgruppe entwickelt computergestützte Methoden, um molekulare Variationen zu analysieren und ihre Auswirkungen auf phänotypische Merkmale besser zu verstehen.

Unsere Forschung
Wie beeinflusst unsere Genetik äußere Merkmale und wie verursacht sie Krebs? Wie werden genetische Faktoren und Umweltfaktoren auf verschiedenen molekularen Ebenen integriert, und wie variabel sind molekulare Zustände zwischen einzelnen Zellen?
Wir nutzen statistische Inferenz und maschinelles Lernen als unsere Hauptwerkzeuge, um diese wissenschaftlichen Fragen zu beantworten. Wachsende Stichprobengrößen und technologische Fortschritte erfordern neue analytische Strategien und Werkzeuge, die auf Datensätze mit Millionen von Beobachtungen skalieren und räumliche sowie zeitliche Abhängigkeiten berücksichtigen. Wir entwickeln grundlegende Methoden in Statistik und maschinellem Lernen, darunter effiziente Parameterinferenz in Modellen zur Untersuchung genetischer Assoziationen und Methoden zur Dimensionsreduktion.
Erfahren Sie mehr über unsere Forschungsgruppe unter https://steglelab.org/
Forschungsthemen
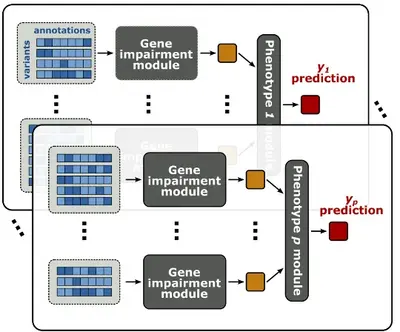
Wir entwickeln statistische Methoden, um häufig übersehene Dimensionen der Genotyp-Phänotyp-Beziehung zu analysieren, einschließlich der genetischen Auswirkungen seltener Varianten und indirekter genetischer Effekte.
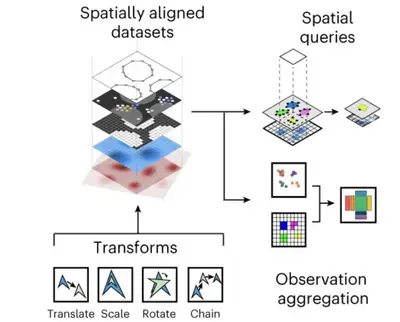
Wir entwickeln grundlegende Methoden, um hochdimensionale molekulare Profile zu integrieren, die in Bulk-Analysen oder auf Einzelzellebene erfasst werden. Dazu gehören Innovationen zur Integration von Multi-Omik-Daten über Zeit und Raum sowie Methoden für räumliche Omik-Technologien.
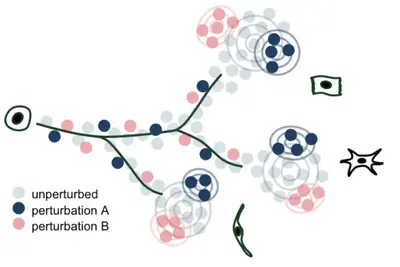
Wir entwickeln Methoden zur Modellierung der Auswirkungen genetischer Perturbationen und zur Ableitung von Kausalitäten. Dabei nutzen wir die Möglichkeiten von gezielten genetischen Perturbationen mithilfe von CRISPR-Screens in Kombination mit Einzelzell-Daten.
Team
Wir sind ein interdisziplinäres und internationales Team, das gemeinsam am Deutschen Krebsforschungszentrum (DKFZ) und dem Europäischen Laboratorium für Molekularbiologie (EMBL) in Heidelberg angesiedelt ist. In unserem Team fördern wir Offenheit und Zusammenarbeit und legen Wert auf Freundlichkeit in allem, was wir tun. Wir sind stolz auf jedes einzelne Mitglied und dessen Leistungen und setzen uns mit Begeisterung für die wissenschaftliche Weiterentwicklung unseres Teams ein.
Offene Stellen
Wir sind stets auf der Suche nach neuen Talenten, um unser Team zu verstärken!
Wenn Sie sich für eine Postdoktoranden oder Doktorandenstelle in unserer Gruppe bewerben möchten, verweisen wir Sie auf das DKFZ Postdoctoral Fellowship und das DKFZ International PhD Programme.
Sollten Sie sich nicht über eine der oben genannten spezifischen Ausschreibungen bewerben, können Sie Ihren Lebenslauf, ein Motivationsschreiben und idealerweise 2 Referenzen in einem einzigen PDF direkt an Oliver Stegle senden.
Wir freuen uns außerdem, Ihnen die Möglichkeit zu bieten, Ihre Bachelor- oder Masterarbeit oder ein Praktikum in unserem Labor durchzuführen. Wenn Sie Interesse haben, kontaktieren Sie uns bitte mit einem Lebenslauf und einem auf die Interessen unseres Labors zugeschnittenen Motivationsschreiben.

Ausgewählte Publikationen
Clarke, B., Holtkamp, E., Öztürk, H., Mück, M., Wahlberg, M., Meyer, K., Munzlinger, F., Brechtmann, F., Hölzlwimmer, F. R., Lindner, J., Chen, Z., Gagneur, J., & Stegle, O.
Marconato, L., Palla, G. , Yamauchi, K. A. , Virshup, I., Heidari, E., Treis, T., Toth, M., Shrestha, R. B., Vöhringer, H., Huber, W., Gerstung, M., Moore, J., Theis, F. J. , & Stegle, O.
Kleshchevnikov, V., Shmatko, A., Dann, E., Aivazidis, A., King, H. W., Li, T., Elmentaite, R., Lomakin, A., Kedlian, V., Gayoso, A., Jain, M. S., Park, J. S., Ramona, L., Tuck, E., Arutyunyan, A., Vento-Tormo, R., Gerstung, M., James, L., Stegle, O., Bayraktar, O. A.
Velten, B., Braunger, J.M., Argelaguet, R., Arnol, D., Wirbel, J., Bredikhin, D., Zeller, G., Stegle, O
Jerber, J., Seaton, D. D., Cuomo, A. S. E., Kumasaka, N., Haldane, J., Steer, J., Patel, M., Pearce, D., Andersson, M., Bonder, M. J., Mountjoy, E., Ghoussaini, M., Lancaster, M. A., Marioni, J. C., Merkle, F. T., Gaffney, D. J., & Stegle, O.
Bonder, M. J., Smail, C., Gloudemans, M. J., Frésard, L., Jakubosky, D., D’Antonio, M., Li, X., Ferraro, N. M., Carcamo-Orive, I., Mirauta, B., Seaton, D. D., Cai, N., Vakili, D., Horta, D., Zhao, C., Zastrow, D. B., Bonner, D. E., Wheeler, M. T., Kilpinen, H., … , Stegle, O.
Kontaktieren Sie uns






