Immundiversität
- Immunologie, Infektion und Krebs

Prof. Dr. Nina Papavasiliou
Abteilungsleitung
Im Gegensatz zu den meisten anderen Zellen unseres Körpers verhalten sich die Zellen des Immunsystems nicht statisch. Sie bewegen sich innerhalb unterschiedlicher Umgebungen und müssen sich dabei ständig an ihr jeweiliges Umfeld anpassen. Diese Anpassungsfähigkeit basiert auf molekularen Mechanismen, die eine einzigartige Diversität hervorbringen. Ziel unserer Forschung ist es, die molekularen Prozesse hinter dieser phänotypischen Diversität der Immunzellen zu verstehen.
Unsere Forschung
Im Gegensatz zu den meisten anderen menschlichen Zellen, bewegen sich die Zellen des Immunsystems fortlaufend in den unterschiedlichsten Körperregionen und müssen sich daher auch ständig entsprechend neu anpassen. Diese Anpassungsfähigkeit beruht auf molekularen Mechanismen die eine solche Informationsvielfalt erzeugen, dass keine Immunzelle der anderen gleicht. Die Zellen des adaptiven Immunsystems (wie B- und T-Lymphozyten) unterscheiden sich jeweils genetisch von ihren Geschwistern, während sich bei den Zellen des angeborenen Immunsystems (wie z.B. Makrophagen) das Transkriptom unterscheiden. Insgesamt zielt unsere Forschung darauf ab, die molekularen Prozesse hinter dieser phänotypischen Vielfalt und somit auch die Grundlagen dieser Anpassungsfähigkeit zu verstehen.
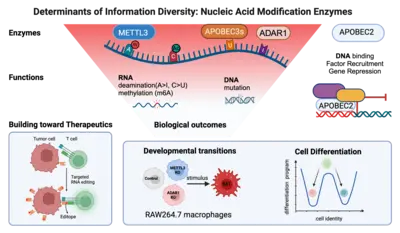
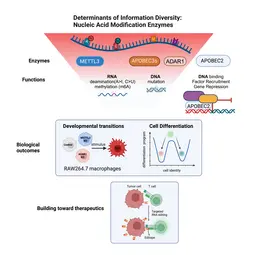
Aus der großen Zahl von Enzymen, von denen bekannt ist, dass sie RNA- oder DNA-Basen durch Hinzufügen (oder Entfernen) bestimmter chemischer Gruppen verändern, haben wir drei Klassen ausgewählt.
(1) Wir untersuchen AID/APOBEC-Proteine, die in der RNA (oder DNA) Cytosin (C) zu Uridin (U) deaminieren, um Uracil (U) zu erhalten. Manche Mitglieder dieser Proteinfamilie sind hochgradig substratspezifisch (AID mutiert z.B. nur DNA); andere eher unspezifisch (APOBEC1 deaminiert z.B. sowohl RNA als auch DNA). Wieder andere wie APOBEC2 binden scheinbar nur DNA – lösen aber trotz geeignetem katalytischen Zentrum keine Deaminierung aus.
(2) Wir analysieren ADAR-Proteine, die in der RNA Adenosin (A) zu Inosin (I) deaminieren. Dadurch wollen wir ihren Beitrag zur Informationsvielfalt in Zellen verstehen und erfahren, warum die Deregulierung dieser Proteine in fast allen untersuchten Gewebetypen zur Onkogenese führt.
(3) Wir beginnen auch mit der Untersuchung von Enzymen, die an der RNA-Methylierung beteiligt sind. Wir möchten das Wechselspiel zwischen Methylierung und Deaminierung anhand derselben Base (z.B. Adenosin) verstehen und herausfinden, welche Rolle es für die Diversität von Immunzellen spielt.
Im Rahmen eines Bottom-up-Ansatzes untersuchen wir die molekularen Mechanismen und biologischen Wirkungen dieser Enzyme auf zellulärer Ebene. So untersuchen wir beispielsweise, wie sich der Verlust einzelner RNA-Modifikationsenzyme auf die zelluläre Differenzierung auswirkt und wie die Überexpression von Enzymen zur Onkogenese führen kann. Langfristig möchten wir verstehen, in welcher Form eine Reihe von Modifikationen das Schicksal eines einzelnen Transkripts bestimmen (seine Lage, seine Halbwertszeit und seine Translationseffizienz). Wir arbeiten dabei auch mit Mathematikern zusammen, um neue Algorithmen dafür zu entwickeln. Schließlich behalten wir immer auch die Translation unserer Forschungsergebnisse im Auge. Wir nutzen zum Beispiel (a) gezieltes RNA-Editing (mittels endogener ADAR) um HLA-präsentierende Zellen in Neoepitope (oder „Editope“) umzuwandeln und dadurch die Immunerkennung (z.B. von Tumoren) zu verstärken. Außerdem (b) arbeiten wir mit Kooperationspartnern an der Entwicklung neuartiger fusogener Proteine, die als Transportpartikel zum gezielten Austausch mit ganz bestimmten Zellen dienen.
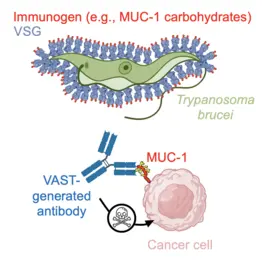
Wir untersuchen Zellvielfalt auch auf Protein-Ebene. Dabei nutzen wir einen einzelligen Parasiten mit einer einzigartigen Fähigkeit als Modell-Organismus – Trypanosoma brucei, den Erreger der afrikanischen Schlafkrankheit. Die Hülle von Trypanosoma brucei ist extrem dicht besetzt von Millionen Kopien eines veränderlichen Proteins (dem „Variant Surface Glycoprotein“, oder VSG) und erzeugt damit eine starke, fokussierte Immunantwort. Der Parasit selbst entzieht sich der dadurch ausgelösten Antikörperreaktion des Immunsystems allerdings sehr effektiv, in dem er seine Proteinhülle einfach „erneuert“ (indem das VSG oder Teile davon verändert werden). Wir untersuchen in unserer Abteilung die biophysikalischen Merkmale und Funktionsweisen dieser veränderlichen Proteinhülle und setzen dabei Antikörper-Repertoire-Analysen und strukturelle Untersuchungsmethoden ein. Unser Ziel besteht darin, die Mechanismen und Wechselwirkungen zwischen der Antikörperreaktion und der als antigene Struktur wirkenden Proteinhülle zu verstehen, die sich jeweils verändern, aneinander anpassen und aufeinander reagieren, und die sich im Laufe ihrer Evolution ständig gegenseitig beeinflussen. Gleichzeitig nutzen wir die ersten Erkenntnisse die wir aus diesem System gewonnen haben, um bessere Immunogene (VAST) mit hohem Translationspotenzial zu entwickeln.
Forschungsteams
Team “RNA Editierung und Modifikationen”
Teamleiter: Dr. Riccardo Pecori
Teammitglieder: Annette Arnold, Dr. Beatrice Casati, Dr. Salvatore Di Giorgio, Valerie Griesche, Eliana Patricelli, Laura Pezzella
Beschreibung der Forschung
RNA-Moleküle durchlaufen verschiedene Verarbeitungsprozesse, die genomische Information diversifizieren, einschließlich Modifikationen einzelner Basen. Eine Fehlregulierung dieser Prozesse steht im Zusammenhang mit zahlreichen Krankheiten.
Unser Team „RNA Editing and Modifications“ untersucht diese Vorgänge. Unser derzeitiger Schwerpunkt liegt auf der RNA-Desaminierung durch die Enzyme ADAR1 und APOBEC1 sowie auf der RNA-Methylierung durch METTL3. In unserer Arbeit erforschen wir die Rolle dieser Enzyme (und der von ihnen katalysierten Modifikationen) in Physiologie und Pathophysiologie mit besonderem Schwerpunkt auf dem Immunsystem.
Wir untersuchen zum Beispiel, wie die Adenosin-Desaminierung die Entwicklungsprozesse von Makrophagen beeinflusst und wie sich der Verlust von ADAR1 auf die Adenosin-Methylierung (m6A-Level) auswirkt. Wir erforschen dies auf der Sequenzebene (unter Verwendung neuartiger Nanopore-Sequenzierungspipelines in Zusammenarbeit mit Terry Lyons in Oxford und Tessy Papavasiliou in Warwick), auf der Ebene der Transkriptverarbeitung (in Zusammenarbeit mit Georg Stoecklin an der Universität Heidelberg) und auf der Ebene der Zelle und der Entscheidungen, die sie treffen kann und die vom richtigen „Lesen“ dieser Modifikationen abhängen.
Wir untersuchen auch die ADAR1 vermittelte Desaminierung, insbesonders, wie ADAR1 durch spezialisierte Guide-RNAs (ADAR1-Engager) rekrutiert werden kann, um Veränderungen in bestimmten, für uns interessanten Transkripten zu erzeugen. Ein aktuelles Beispiel ist der Einsatz neuartiger Präzisionswerkzeuge, mit denen endogenes ADAR1 mit hoher Effizienz in Regionen von Transkripten rekrutiert wird, die für HLA-präsentierte Peptide kodieren, und diese Peptide so verändert werden, dass sie immunogen werden (d. h. von T-Zellen „entdeckt“ werden können). Dieser Ansatz birgt ein beträchtliches therapeutisches Potenzial, das wir von der mechanistischen Ebene (Generierung neuartiger Guide-RNAs) bis hin zur Ebene des Organismus (präklinische Studien zur Tumorabstoßung nach Verabreichung von ADAR1-Engagern) nutzen.
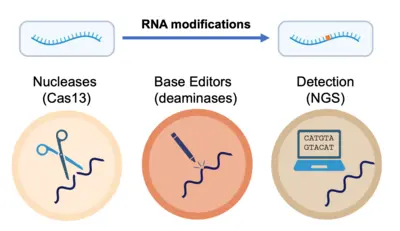
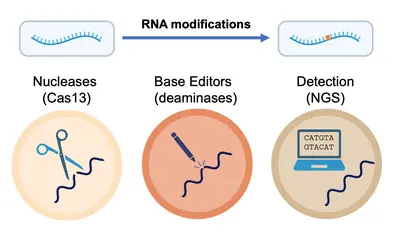
Im weiteren Sinne nutzen wir das Wissen, das wir bei der Untersuchung von RNA-Modifikationen im Kontext der Zelle gewonnen haben, zur Entwicklung von Spitzentechnologien für den präzisen Nachweis und die Manipulation von RNA auf Ebene einzelner Nukleotide. Gemeinsam entwickeln und implementieren wir fortschrittliche Strategien für den schnellen und genauen Nachweis und die Quantifizierung von RNA-Modifikationen in verschiedenen Datentypen, um ein tieferes Verständnis der Onkogenese und der Immunreaktionen zu erlangen, aber auch den Weg für neue therapeutische Strategien zu ebnen.
Förderungen
- 2022, DFG GRK2727 "ImCheck" (Projekt A1.2)
- 2022, Deutsche Forschungsgemeinschaft (DFG, German Research Foundation) project number 439669440 TRR319 RMaP TP 04 awarded to F.N.P.
- 2021, HI-TRON Kick-Start Seed Funding Program 2021 awarded to Dr. Pecori and Prof. Dr. Tenzer (University Medical Center Mainz).
- 2020, Ministry of Science, Research and the Arts of Baden-Württemberg for COVID-19 research (grant agreement no. Kap. 1499 TG 93 to Dr. Pecori, Prof. Dr. Papavasiliou and Prof. Dr. Miethke (Medical Faculty of Mannheim)).
- 2020, European Research Council award (ERC PoC "EpiMODkit") to Prof. Dr. Papavasiliou.
Partnerschaften
- Prof. Dr. med. Michael Platten (DKFZ)
- Prof. Dr. Rienk Offringa (DKFZ)
- Dr. Rafael Carretero (DKFZ)
- Prof. Dr. Mark Helm (Uni Mainz)
- Prof. Dr. Rocio Sotillo (DKFZ)
- Prof. Dr. Georg Stoecklin (Uni Heidelberg)
- Prof. Dr. Stefan Tenzer (University Medical Center Mainz)
- Prof. Dr. Terry Lyons (University of Oxford)
- Prof. Dr. Tessy Papavasiliou (University of Warwick)
- Prof. Dr. Thomas Miethke (Medical Faculty of Mannheim)
- Prof. Dr. Thorsten Stafforst (University Hospital Tübingen)
- Prof. Dr. Qiang Pan-Hammarström (Karolinska Institutet)
Publikationen
- Latifi, et al. Precise and efficient C-to-U RNA base editing with SNAP-CDAR-S. Nucleic Acids Research, 2023.
- Casati, et al. Tumor warm-up: RNA editing boosts tumor immunogenicity. BioRxiv, 2023.
- Pecori, et al. ADAR1-mediated RNA editing promotes B cell lymphomagenesis. Science, 2023.
- Pecori, et al. ADAR RNA editing on antisense RNAs results in apparent U-to-C base changes on overlapping sense transcripts. Front. Cell Dev. Biol., 2023.
- Casati, et al. Rapid, adaptable and sensitive Cas13-based COVID-19 diagnostics using ADESSO. Nature Communications, 2022.
- Pecori, et al. Functions and consequences of AID/APOBEC-mediated DNA and RNA deamination. Review, Nature Reviews Genetics, 2022.
- Stroppel, et al. Harnessing self-labeling enzymes for selective and concurrent A-to-I and C-to-U RNA base editing. Nucleic Acids Research, 2021.
- Kluesner, et al. MultiEditR: The first tool for the detection and quantification of RNA editing from Sanger sequencing demonstrates comparable fidelity to RNA-seq. Molecular Therapy Nucleic Acids, 2021.
- Casati, et al. ADAR-mediated RNA editing and its therapeutic potentials. RNA Technologies, Book Chapter, Springer Series, Vol. 11, 2021.
- Lerner, et al. C-to- U RNA editing: from computational detection to experimental validation. Book Chapter, Methods in Molecular Biology, 2021.
- Pecori, Papavasiliou. It takes two (and some distance) to tango: how ADARs join to edit RNA. News & Views, Nature Structural Molecular Biology, 2020.
- Tasakis, et al. ADAR1 can drive Multiple Myeloma progression by acting both as an RNA editor of specific transcripts and as a DNA mutator of their cognate genes. BioRxiv, 2020.
- Lerner, et al. RNA Editors, Cofactors, and mRNA Targets: An Overview of the C-to-U RNA Editing Machinery and Its Implication in Human Disease. Review, Genes, 2019.
Team “Desaminasen und Lymphozytenschicksalbestimmung”
Teamleiter: Dr. Elias Amro
Teammitglieder: Christos Gkougkousis, Nina Lemmen
Beschreibung der Forschung
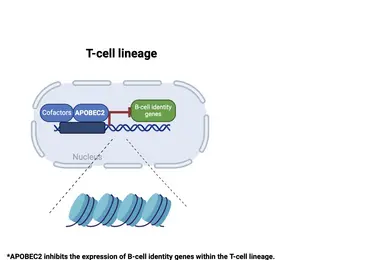
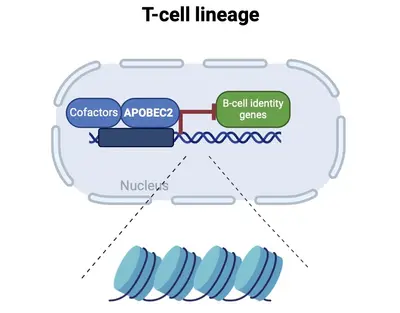
Die Differenzierung in spezifische Zellen und die Aufrechterhaltung der Zellidentität erfordern molekulare Programme, die von Transkriptionsfaktoren gesteuert werden, die nicht nur zellspezifische Identitäten aufrechterhalten, sondern auch alternative Zelldifferenzierungswege unterdrücken. Unsere jüngsten Forschungsarbeiten konzentrieren sich auf die Erforschung welche Rolle APOBEC2 bei der Spezifizierung der lymphoiden Abstammungslinien spielt. APOBEC2 gehört zur hochkonservierten Familie der Apolipoprotein B mRNA-editing enzyme catalytic polypeptide-like (APOBEC), die für ihre zinkabhängige Deaminase-Aktivität bekannt ist (Pecori et al., 2022; Wedekind et al., 2003; Harris et al., 2002; Powell et al., 2014). Während die meisten Mitglieder dieser Familie die Umwandlung von Cytidin in Uridin innerhalb der DNA oder RNA katalysieren, fehlt APOBEC2 diese enzymatische Funktion. Trotzdem spielt APOBEC2 eine unkonventionelle Rolle bei der Regulierung biologischer Funktionen sowohl in vitro als auch in vivo. Bei Mäusen führt die Deletion von Apobec2 zu Muskelschwäche, reduzierter Muskelmasse und einer Verschiebung von schnell zu langsam zuckender Muskelfaserbildung (Sato et al., 2010; Mikl et al., 2005; Sato et al., 2018). Apobec2 wurde durch Mutationen und veränderte Genexpression auch mit der Krebsentwicklung in Verbindung gebracht (Okuyama et al., 2012; Wei et al., 2024; Li et al., 2019). Jüngste Forschungsarbeiten unseres Labors (Lorenzo, Molla, Amro et al., 2024) haben eine neue Rolle für APOBEC2 als Transkriptionsfaktor aufgedeckt, der die Muskelidentität aufrechterhält, indem er Nicht-Muskel-Gene unterdrückt. Über die Muskelbiologie hinaus zeigt unsere aktuelle Arbeit, dass ein APOBEC2-Mangel bei Mäusen und Menschen zu Defekten bei der Spezifizierung der lymphatischen Zelllinien führt. Menschliche Individuen mit homozygoten APOBEC2-Mutationen zeigen geschwächte Immunantworten auf Impfungen und eine erhöhte Anfälligkeit für Immunstörungen (Amro, Gkougkousis et al., unveröffentlicht). Um die Rolle und Funktion von APOBEC2 in Gesundheit und Krankheit weiter zu entschlüsseln, konzentriert sich die Forschung unseres Teams auf zwei Hauptziele:
- das Verständnis der molekularen Mechanismen, durch die Apobec2 die Spezifizierung der lymphoiden Abstammung reguliert;
- die Aufklärung der Auswirkungen von Apobec2-Mutationen auf die Funktionen des menschlichen Immunsystems.
Partnerschaften
- Dr. Charles Imbusch (Uni Mainz)
- Prof. Dr. Christoph Dieterich (Uni Heidelberg)
- Prof. Dr. Lennart Hammarström (Karolinska Institutet)
- Prof. Dr. Qian Pan Hammarström (Karolinska Institutet)
- Prof. Dr. Fowzan AlKuraya (King Faisal Hospital, Riyadh)
Publikationen
- Lorenzo, Molla, Amro, et al. APOBEC2 safeguards skeletal muscle cell fate through binding chromatin and regulating transcription of non-muscle genes during myoblast differentiation. Proceedings of the National Academy of Science, 2024.
- Pecori, et al. Functions and consequences of AID/APOBEC-mediated DNA and RNA deamination. Nature Reviews Genetics, 2022.
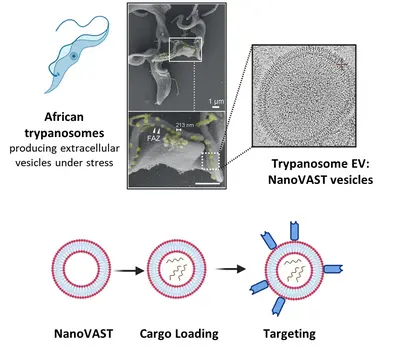
Team “T. brucei-abgeleitete Instrumente: NanoVAST”
Teamleiter: Dr. Katerina Spyridopoulou
Teammitglieder: Laura Pezzella, Niklas Veocic
Beschreibung der Forschung
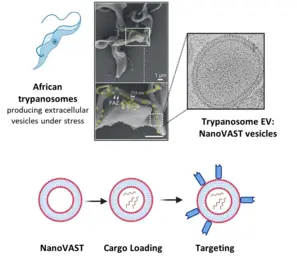
Die gezielte Verabreichung von therapeutischen Wirkstoffen an bestimmte Zelltypen ist nach wie vor eine große Herausforderung in sich entwickelnden Forschungs-bereichen wie der Gentherapie, der RNA-Modifikation und der Verabreichung von Medikamenten. Technologien wie CRISPR/Cas bergen zwar ein immenses therapeutisches Potenzial, ihre klinische Umsetzung wird jedoch vor allem durch den Mangel an sicheren, effizienten und zielgerichteten Verabreichungssystemen behindert. Die derzeitigen Verabreichungsmethoden, einschließlich Elektroporation und virale Vektoren, leiden unter Einschränkungen wie Toxizität und Immunogenität.
Um das Problem zu lösen, setzt unser Team nanoVAST ein, eine neuartige Plattform, die von unseren Kollegen der Panosome GmbH entwickelt wurde. NanoVAST besteht aus einer patentierten vesikulären Phospholipid-Doppelschicht, die auf der skalierbaren Produktion von extrazellulären Nanovesikeln, gewonnenaus afrikanischen Trypanosomen, basiert. Da nanoVAST biogen ist, weist es keine Toxizität auf, während seine fusogenen Eigenschaften die zytoplasmatische Anlieferung seiner Ladung ermöglichen. Entscheidend ist, dass es sehr zielgerichtet ist, da seine Oberfläche mit einer spezifischen, auf Sortase basierenden Chemie - genannt VAST - modifiziert werden kann, um zielgerichtete Komponenten anzubringen.
Wir optimieren nanoVAST für die In-vitro-, Ex-vivo- und In-vivo-Verabreichung von therapeutischen Ladungen und erforschen seine biochemischen Eigenschaften, um hochspezifische Strategien für die Ausrichtung auf Zellen zu entwickeln. Unsere Arbeit konzentriert sich auf die Entwicklung von ladungsspezifischen Beladungsmethoden und zellspezifischen Targeting-Ansätzen für Krebs- und Immunzellen (wie T-Zellen und NK-Zellen). Zu den Frachttypen, die derzeit untersucht werden, gehören TCR-mRNA, CAR-mRNA, siRNA, Leit-RNAs für RNA-Editierung und DNA-Vektoren. Wir entwickeln und verwenden auch modernste Technologien, um die optimierte Produktion von nanoVAST, die Beladung mit Ladung und die effektive und funktionelle Verabreichung an die Zellen zu bewerten.
Durch die Verbesserung der gezielten Verabreichung von Arzneimitteln und Gentherapiesystemen hat diese Technologie das Potenzial, neue therapeutische Wege für zahlreiche Krankheiten zu eröffnen, für die es derzeit keine wirksamen Behandlungen gibt.
Förderungen
- European Research Council 2022 Proof of Concept Grant (nanoVAST)
- DKFZ seed funding program 2024: Kick-start DreamTeam Grant
Partnerschaften
- Prof. Dr. med. Michael Platten (DKFZ)
- Prof. Dr. Joachim Spatz (MPI for Medical Research)
- Synthetic Immunology Research Network
Publikationen
- Hempelmann, et al. Nanobody-mediated macromolecular crowding induces membrane fission and remodeling in the African trypanosome. Cell Reports, 2021.
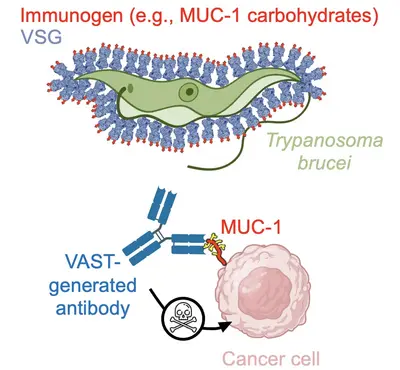
Team “T. brucei-abgeleitete Instrumente: VAST und die Entwicklung von Antikörpern als Therapeutika”
Teamleiter: Dr. Joseph (Joey) Peter Verdi
Teammitglieder: Dr. Monica Chandra, Dr. Anastasia Gkeka, Dr. Nataliia Kashpur, Seneca Kinn-Gurzo, Eblina Kelmendi, Dr. Jose Paulo Lorenzo, Dr. Colin Miller, Eleftheria Papamanoglu, Sandra Ruf, Dr. Eirini Sidiropoulou, Dr. Katharina Urban
Beschreibung der Forschung
Keytruda, Humira, Herceptin… Die Behandlung zahlreicher menschlicher Krankheiten mit antikörperbasierten Therapien ist heute eine Erfolgsgeschichte. Unser Team hat es sich zum Ziel gesetzt, das Spektrum der mit Antikörpern adressierbaren Erkrankungen mithilfe unserer intern entwickelten Technologieplattform zur Antikörpergenerierung zu erweitern. Während hochqualitative Antikörper gegen Proteinantigene mittlerweile vergleichsweise einfach herzustellen sind, bleibt die reproduzierbare Erzeugung gleichwertiger Antikörper gegen kleine Moleküle und Kohlenhydrate eine ungelöste Herausforderung. Dabei sind diese kleinen Moleküle genauso eng mit Krankheiten verknüpft oder sogar deren Ursache wie Proteine – dennoch wurde dieses Feld bislang nicht überzeugend erschlossen. Genau hier setzen wir an: Wir fokussieren uns gezielt auf diese „schwierigen Targets“ mit dem Ziel, innovative Therapien zu entwickeln.
Um diese Herausforderungen zu meistern, haben wir eine Plattformtechnologie entwickelt, die auf der einzigartigen Oberflächenarchitektur des afrikanischen Trypanosoms basiert. Die Oberfläche von Trypanosoma brucei ist extrem dicht mit 10 Millionen Kopien eines Proteins – dem variablen Oberflächenglykoprotein (VSG) – besetzt. Diese außergewöhnlich dichte und präzise ausgerichtete Anordnung von Antigenen stimuliert das Immunsystem auf bemerkenswerte Weise, indem sie die krankheitsrelevante Antikörperreaktion gezielt auf Epitope lenkt, die ausschließlich an der Spitze des VSG-Proteins liegen. Dieser sogenannte „Epitopfokussierungseffekt“ gilt als einer der stärksten seiner Art in der Natur. Unser Team hat sich diesen Mechanismus zunutze gemacht, indem es schwer zugängliche Zielstrukturen auf der Oberfläche von VSG fusionierte – und so das VSG Immunogen Array by Sortase Tagging (VAST) entwickelte.
Die Anwendbarkeit der VAST-Technologie konnte bereits eindrucksvoll demonstriert werden: So wurden Antikörper mit pico- und femtomolarer Affinität gegen die Opioide Fentanyl und Carfentanil generiert, die in Tiermodellen eine vollständige Prävention der Opioid-Toxizität ermöglichen. Ebenso gelang die Herstellung hochwertiger Antikörper gegen Kohlenhydrate auf Mucin 1 (MUC1), einem vielversprechenden Ziel bei soliden Tumoren.
Diese bahnbrechende Technologie, ihre innovativen Antikörperprodukte und jedes außergewöhnlich talentierte Mitglied unseres Teams bilden das Fundament der Panosome GmbH, einem Biotechnologie-Start-up mit Sitz in der Nähe von Heidelberg.
Förderungen
- 2024, KMU-innovativ (BMBF) project “CarboVAST” awarded to Panosome GmbH with DKFZ as a collaborating institution (Dr. Verdi and Prof. Papavasiliou as project leaders)
- 2024, KMU-innovativ (BMBF) project “GliomAb” awarded to Panosome GmbH with DKFZ as a collaborating institution (Dr. Verdi and Prof. Papavasiliou as project leaders)
- European Research Council 2020 Proof of Concept Grant (EpiMODkit)
Partnerschaften
- Prof. Dr. Rienk Offringa (DKFZ)
- Prof. Dr. med. Michael Platten (DKFZ)
- Dr. Aubry Miller (DKFZ)
- Dr. Erec Stebbins (DKFZ)
Publikationen
- Urban and Gkeka et al., The fentanyl-specific antibody FenAb024 can shield against carfentanil effects. Toxicology Letters, 2024.
- Triller et al., A trypanosome-derived immunotherapeutics platform elicits potent high-affinity antibodies, negating the effects of the synthetic opioid fentanyl. Cell Reports, 2023.
- Gkeka and Aresta-Branco et al., Immunodominant surface epitopes power immune evasion in the African trypanosome. Cell Reports, 2023.
- Triller et al., Immunization with Genetically Modified Trypanosomes Provides Protection against Transmissible Spongiform Encephalopathies. International Journal of Molecular Sciences, 2022.
Mitarbeiter
-

Prof. Dr. Nina Papavasiliou
Abteilungsleitung
-

Dr. Elias Amro
Teamleiter "Desaminasen und Lymphozytenschicksalbestimmung"
- Profil anzeigen

Christoph Tim Pufall
Administrative Assistant
-
Annette Arnold
Technische Assistentin
-
Dr. Monica Chandra
Postdoctoral researcher
-
Dr. Salvatore Di Giorgio
Postdoctoral researcher
-
Anastasia Gkeka
Postdoctoral researcher
-
Christos Gkougkousis
Doctoral researcher
-

Valerie Griesche
Doctoral researcher
-
Nataliia Kashpur
-
Seneca Kinn-Gurzo
Doctoral researcher
-
Dr. Jose Paulo Lorenzo
Postdoctoral researcher
-
Eleftheria Papamanoglou
-

Dr. Riccardo Pecori
Stellvertretende Leitung und Teamtleiter "RNA Editierung und Modifikationen"
-
Laura Pezzella
Doctoral researcher
-
Sandra Ruf
-
Dr. Eirini Sidiropoulou
Postdoctoral researcher
-

Dr. Aikaterini Spyridopoulou
Teamleiterin "T. brucei-abgeleitete Instrumente: NanoVAST"
-
Dr. Joseph Peter Verdi
Teamleiter "T. brucei-abgeleitete Instrumente: VAST und die Entwicklung von Antikörpern als Therapeutika"
-

Colin Gregory Miller
Postdoctoral researcher
-
Dr. Katharina Urban
-

Eblina Boddin
Alumni
Menschen kommen und gehen in der Wissenschaft. Das ist auch Teil der Forschung. In unserem Team auch haben viele Leute seit der Gründung der Forschungsgruppe im Jahr 2016 zusammengearbeitet. Sie alle haben geholfen, in unseren Foschungsprojekten wichtige Forrtschritte zu machen. Hier sind alle Personen aufgelistet, die mit uns gearbeitet haben (in Klammern die ehemalige Funktion innerhalb unserer Gruppe).
Ausgewählte Publikationen
Lorenzo JP, Molla L, Amro EM, Ibarra IL, Ruf S, Neber C, Gkougkousis C, Ridani J, Subramani PG, Boulais J, Harjanto D, Vonica A, Di Noia JM, Dieterich C, Zaugg JB, Papavasiliou FN.
Pecori R, Rent W, Pirmoradian M, Wang X, Liu D, Berglund M, Li W, Tasakis RN, Di Giorgio S, Ye X, Li X, Arnold A, Wüst S, Schneider M, Selvasaravanan K, Fuell Y, Stafforst T, Amini RM, Sonnevi K, Enblad G, Sander B, Wahlin BE, Wu K, Zhang H, Helm D, Binder M, Papavasiliou FN.
Gkeka A, Aresta-Branco F, Triller G, Vlachou EP, van Straaten M, Lilic M, Olinares PDB, Perez K, Chait BT, Blatnik R, Ruppert T, Verdi JP, Stebbins CE, Papavasiliou FN.
Triller G, Vlachou EP, Hashemi H, van Straaten M, Zeelen JP, Kelemen Y, Baehr C, Marker CL, Ruf S, Svirina A, Chandra M, Urban K, Gkeka A, Kruse S, Baumann A, Miller AK, Bartel M, Pravetoni M, Stebbins CE, Papavasiliou FN, Verdi JP.
Casati B, Verdi JP, Hempelmann A, Kittel M, Gutierrez Klaebisch A, Meister B, Welker S, Asthana S, Di Giorgio S, Boskovic P, Man KH, Schopp M, Ginno PA, Radlwimmer B, Stebbins CE, Miethke T, Papavasiliou FN, Pecori R.
Pecori R, Di Giorgio S, Lorenzo JP, Papavasiliou FN.
Hempelmann A, Hartleb L, van Straaten M, Hashemi H, Zeelen JP, Bongers K, Papavasiliou FN, Engstler M, Stebbins CE, Jones NG.
Casati B, Stamkopoulou D, Tasakis RN, Pecori R.
Kluesner M, Tasakis RN, Lerner T, Arnold A, Wüst S, Binder M, Webber BR, Moriarity BS, Pecori R.
Kontaktieren Sie uns