Abteilung Neuroblastom-Genomik
PD Dr. Frank Westermann
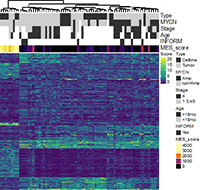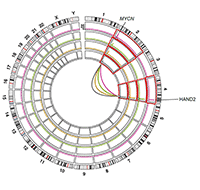
Das Neuroblastom ist eine Krebsart, die ausschließlich bei Kindern auftritt und sich besonders vielseitig verhält. Es entsteht in den ersten Lebensjahren aus nicht ausgereiften Zellen des peripheren Nervensystems. Ungewöhnlich oft bilden sich Neuroblastome von selbst zurück. Sie können aber auch sehr aggressiv fortschreiten und zum Tod führen. Neuroblastome, die einen Mechanismus zur Telomerverlängerung aufweisen, haben in der Regel eine schlechte Prognose, wohingegen niedriggradige Tumoren meist keinen solchen Mechanismus aufweisen. Unterschiedliche Neuroblastom-Subtypen können die Telomere entweder durch Aktivierung des Enzyms Telomerase (als Folge von MYCN-Aktivierung oder TERT-rearrangement) oder durch den alternative lengthening of telomeres (ALT) Mechanismus verlängern.
Das übergeordnete Ziel unserer Arbeiten ist eine möglichst genaue individuelle Risikoabschätzung mittels molekularer Marker bereits zum Zeitpunkt der Diagnose, um bei jedem Patienten die Therapieintensität besser an die Eigenschaften des Tumors anzupassen. Des Weiteren arbeiten wir an der Entwicklung von maßgeschneiderten Therapien für die aggressiven Verlaufsformen des Neuroblastoms. Hierzu benötigen wir ein genaues Verständnis der Entstehung, Progression und Therapieresistenz von Neuroblastomen.
Spezifisches Ziel unserer Arbeit ist, über eine umfassende molekulargenetische Charakterisierung der unterschiedlichen Neuroblastom-Subtypen (z. B. mittels globaler Genexpressions-, DNA-Methylierungsanalyse und Hochdurchsatzsequenzierung) die ursächlichen genetischen, epigenetischen und metabolischen Veränderungen des Neuroblastoms zu finden und dessen unterschiedlichen klinischen Verlauf aufzuklären. Desweiteren arbeiten wir darauf hin, die regulatorischen Netzwerke, die das Epigenom und Transkriptom von Neuroblastom Zellen definieren, besser zu verstehen und ihre Rolle in der Tumorentstehung, Rezidivbildung und Metastasierung zu entschlüsseln. Durch eine Kombination aus Ganzgenomsequenzierung, Genexpressionsanalysen, Histon-Modifikations-Analysen (ChIP-seq) und 3D-Chromatin-Kontext-Analysen (z.B. Circular chromatin conformation capturing, 4C oder HiC), charakterisieren wir chromosomale Umlagerungen in Neuroblastomzellen, die regulative genomische Bereiche (sogenannte „Super Enhancer“) in den Kontext von Onkogenen translozieren und diese dadurch aktivieren. Mit Hilfe von Einzelzellanalysen und räumlich aufgelösten Transkriptionsanalysen (spatial transkriptomics) von Neuroblastom-Tumoren und normalen embryonalen und fetalen Geweben verschiedener Entwicklungsstufen des peripheren Nervensystems untersuchen wir die entwicklungsbiologischen Ursprünge und Entwicklungsverläufe des Neuroblastoms. Unsere (epi)genetischen Arbeiten ergänzen wir durch die Analyse der metabolischen Netzwerke in Neuroblastom-Zellen. Dabei sind wir besonders an der Frage interessiert, ob diese Netzwerke einen Angriffspunkt für neue Therapien darstellen.
Um spezifische Schwachstellen von Neuroblastom-Zellen auszumachen und systematisch Zielstrukturen für die personalisierte Medizin zu identifizieren, nutzen wir funktionelle Hochdurchsatzanalysen (siRNA-/shRNA-Screening-Verfahren und Wirkstoff-Screening-Verfahren). Wir suchen dabei besonders nach Schwachstellen, die mit spezifischen Veränderungen in den Krebszellen assoziiert sind. Wir haben langjährige Erfahrung in der präklinischen Entwicklung und Validierung von spezifischen Zielstrukturen und arbeiten dabei eng mit Partnern aus der Klinik und Industrie zusammen (z. B. bei der Entwicklung von Proteolysis Targeting Chimeras (PROTACs) für den gezielten Abbau von ungewünschten Proteinen in Krebszellen).
Unsere genetischen und molekularen Hochdurchsatzdaten kombinieren wir mit systembiologischen Ansätzen, um einen Einblick in die genetische Evolution von Neuroblastom-Tumoren zu erhalten und die zugrundeliegenden onkogenen Netzwerke zu verstehen.
Weiterführende Informationen sind nur auf Englisch vorhanden.
NB super-enhancer visualization tool
NB developmental programs visualization tool