Abteilung Proteomik von Stammzellen und Krebs
Prof. Dr. Jeroen Krijgsveld
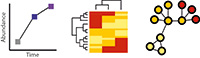
Die Funktionalität von Proteinen lässt sich ableiten aus Proteomik-Experimenten, mit deren Hilfe sich die Proteinexpression im zeitlichen Verlauf (links) oder bei verschiedenen Bedingungen und im Vergleich zu anderen Proteinen (Mitte) bestimmen lässt, auch lassen sich die Protein-Interaktionspartner (rechts) bestimmen.
© dkfz.de
Ziel unserer Forschung ist es, tiefere Erkenntnisse über komplexe Proteome zu gewinnen und Methoden zu entwickeln, mit denen wir herausfinden können, welche zeitlichen und räumlichen Abläufe Proteome dynamisch regulieren. Diese Methoden setzen wir hauptsächlich dazu ein, um Fragestellungen auf den Gebieten der Pluripotenz von Stammzellen sowie der Tumorbiologie zu beantworten.
Der methodische Schwerpunkt unserer Abteilung ist die quantitative Proteomik, unterstützt durch Expertise in den Bereichen Biochemie, analytische Chemie und Bioinformatik. Wir verfügen über hochmoderne Massenspektrometer, mit denen wir Tausende von Proteinen pro Probe analysieren können. Diese Technik haben wir beispielsweise zur Charakterisierung von murinen Blutstammzellen eingesetzt, wodurch wir neue Erkenntnisse über die ersten entscheidenden Abläufe der Bildung neuer Blutzellen in vivo gewinnen konnten. Eine Analyse der zeitlichen Änderungen des Proteoms während der Reprogrammierung von Fibroblasten hin zu induzierten pluripotenten Stammzellen (iPSC) führte zur Identifizierung von Proteinen, die eine Schlüsselrolle während der Entwicklung zur Pluripotenz spielen.
Wir haben in unserem Labor ebenfalls neue Methoden zur Probenvorbereitung entwickelt, die es uns ermöglichen, auch kleinste Proben mit Hilfe eines Robotersystems standardisiert und höchst sensitiv zu analysieren. Diese Technologie ist besonders geeignet für Proben von begrenzter Menge (z.B. klinische Proben) und zur Analyse des Tumorproteoms.
Darüber hinaus haben wir Methoden etabliert, mit denen wir sezernierte Proteine mittels click-chemistry anreichern und proteomisch untersuchen können. Diesen Ansatz nutzen wir, um interzelluläre Kommunikation von Tumorzellen zu untersuchen, sowie zur Entschlüsselung fundamentaler Mechanismen von Sekretionswegen.
Ein weiterer Schwerpunkt unserer Arbeit liegt darin, zu untersuchen, wie Proteine mit DNA und RNA interagieren, und die Funktion dieser Proteine bei der Genomregulierung zu verstehen. In Zusammenarbeit mit Matthias Hentze vom EMBL haben wir beispielsweise eine Methode entwickelt, mit der wir mRNA-Interaktome in vivo analysieren können. Unser Ziel ist es, diese Erkenntnisse auf andere RNA-Arten zu übertragen. Zusätzlich etablieren wir neue Ansätze zur Untersuchung von Protein-Interaktionen im Chromatin. Unser Fokus liegt dabei auf der Analyse der Zusammensetzung und der Dynamik von Proteinnetzwerken zentraler Transkriptionsfaktoren und chromatinmodifizierender Enzyme. Letztendlich versuchen wir so in Kombination mit ChIP-seq und RNA-seq zu verstehen, welche bislang unbekannten Proteine und Mechanismen eine Schlüsselrolle bei der Kontrolle der Genexpression einnehmen und wie dies im Fall von Krebs fehlreguliert ist.
Die weiterführenden Seiten sind derzeit nur auf englisch verfügbar.

