Abteilung Chromatin-Netzwerke
Prof. Dr. Karsten Rippe
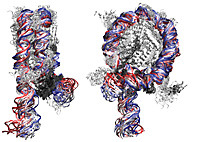
Computersimulation der dynamischen Nukleosomstruktur. Das Nukleosom ist der Grundbaustein des Chromatins und besteht aus einem Histonproteinkern und umwickelter DNA. In einer menschlichen Zelle ist das Genom von sechs Milliarden DNA-Basenpaaren in ca. 30 Millionen Nukleosomen organisiert. Die Abbildung zeigt Computersimulationen der dynamischen Nukleosomenkonformationen über einen Zeitraum von 2 Nanosekunden. Die Histonproteine sind weiß dargestellt und die Farbgebung der DNA entspricht der Simulationszeit (zunehmend von Rot über Weiß zu Blau). Für Details zur Untersuchung von Nukleosom- und Chromosomeigenschaften in Computersimulationen siehe Ettig et al. 2011, Biophys. J. and Kepper et al. 2008, Biophys. J.
© dkfz.de
Die Abteilung "Chromatin-Netzwerke" am DKFZ und BioQuant ist ein interdisziplinäres Team, das Methoden der Molekular- und Zellbiologie mit denen der Physik kombiniert. In den Arbeiten wird der Zusammenhang der dynamischen Organisation des Genoms im eukaryotischen Zellkern mit dem Auslesen, Prozessieren, Erhalten und Transferieren der in der DNA-Sequenz kodierten Information untersucht. Ein besonderer Schwerpunkt liegt auf der Konformation und den dynamischen Eigenschaften von Chromatin. Damit wird der Komplex bezeichnet, den das DNA-Genom mit Histonen und anderen chromosomalen Proteinen bildet. Sowohl die DNA als auch die Proteinkomponente des Chromatins unterliegen verschiedenen posttranslationalen Modifikationen. Dazu gehören DNA- und Histon-Methylierung sowie die Acetylierung und Phosphorylierung von Histonen. Diese epigenetischen Signale bestimmen das Genexpressionsmuster der Zelle und können über die Zellteilung hinaus weitergegeben werden. Die entsprechenden Prozesse sind eng mit der Organisation des Chromatins verküpft, die wiederum von zentraler Bedeutung für den Zugang anderer Proteinfaktoren zur genetischen Information ist. Forschungsziel der Gruppe ist es, in einer integrierten Darstellung abzubilden, wie das dynamische Gleichgewicht zwischen unterschiedlichen Chromatinzuständen die Funktionen des Genoms bestimmt.
Das Verständnis epigenetischer Regulation wird für die Diagnose und Therapie von Krebs, Entwicklungsstörungen und anderer Krankheiten immer wichtiger. In diesem Zusammenhang führt die Arbeitsgruppe Analysen epigenetischer Netzwerke mit biophysikalischen Methoden wie der Fluoreszenzspektroskopie/-mikroskopie in lebenden Zellen durch. Die experimentellen Ergebnisse dienen als Basis für Modellierungsansätze, die auf die Entwicklung quantitativer Modelle der dynamischen Chromatinorganisation abzielen. Komplementär werden genomweite Studien zur Nukleosompositionierung, Proteinbindung und Histonmodifikationen in Zellpopulationen mittels DNA-Sequenzierung durchgeführt. Die Erkenntnisse aus den unterschiedlichen Forschungsansätzen werden in einen systembiologischen Ansatz integriert, um epigenetische Netzwerke zu analysieren. Die Arbeit der Forschungsgruppe ist für die translationale medizinische Erforschung der komplexen Effekte epigenetisch wirksamer Medikamente (z.B. Inhibitoren von Histondeacetylasen oder DNA-Methylasen) in der Krebstherapie von Bedeutung. Deshalb ist geplant, die Untersuchungen immortalisierter humaner und muriner Zelllinien auf Studien primärer Krebszellen zu erweitern.
Externe Bioquant-Webseite

