Pädiatrische Gliomforschung
- Funktionelle und Strukturelle Genomforschung
- KiTZ

Prof. Dr. David Jones
Division Head
Jeder zweite Tumor des Zentralnervensystems im Kindes- und Jugendalter (0-19 Jahre) ist ein Gliom, somit sind die Gliome die häufigste Form der kindlichen Hirntumore. Grundsätzlich werden zwei Kategorien unterschieden: niedriggradige und hochgradige Gliome.
Unsere Forschung
Die niedriggradigen Gliome sind eine histologisch und biologisch sehr vielfältige Sammlung von Tumorentitäten, denen in den meisten Fällen eine genetische Veränderung im mitogen-activated protein kinase (MAPK) Signalweg gemeinsam ist. Aufgrund der hohen Überlebensraten werden sie im Allgemeinen als gutartige Tumoren angesehen. Bedingt durch die Therapien und den Tumor selbst geht die hohe Überlebensrate jedoch häufig auf Kosten der Lebensqualität. Zahlreiche Rückfälle über viele Jahre hinweg sind nicht selten und stellen eine große Belastung für die Patienten und ihre Familien dar. Durch unsere Arbeit möchten wir mehr über den biologischen Hintergrund und das ungewöhnliche Wachstumsverhalten dieser Tumoren lernen mit dem Ziel, neue Therapiemöglichkeiten zu identifizieren, die auf die Biologie der jeweiligen Tumoren zugeschnitten sind und dadurch möglicherweise weniger Nebenwirkungen mit sich bringen.
Im Gegensatz dazu haben die hochgradigen Gliome eine ausgesprochen schlechte Prognose. Diese hoch aggressiven Tumoren, meistens Glioblastome oder diffus intrinsische Ponsgliome (DIPG), zeigen typischerweise eine deutlich höhere genomische Instabilität als nieddriggradige Läsionen. Die kombinierte Störung zahlreicher zellulärer Prozesse und Signalwege bewirkt eine rasche Teilungsrate der Tumorzellen, die das umliegende Hirngewebe diffus infiltrieren. Das hat in aller Regel den Tod des Patienten zufolge. Unser wissenschaftliches Ziel in diesem Bereich ist es, die Tumorheterogenität sowohl innerhalb ein und desselben Tumors als auch zwischen verschiedenen Individuen zu verstehen und dadurch die entscheidenden Veränderungen in der Zellmaschinerie zu erkennen. Wir versuchen dann, diese mit Hilfe von verschiedenen Tumormodellen nachzustellen, um herauszufinden, welchen Beitrag sie jeweils dazu leisten, aus einer Zelle eine Tumorzelle zu machen. Weiterhin werden wir die Modelle nutzen, um neue zielgerichtete Therapien sinnvoll zu testen.
Die gesamte Laborarbeit basiert auf der Anwendung neuester und innovativster genomischer (next-generation DNA/RNA sequencing), epigenetischer (DNA methylation, ChIPseq) und funktioneller (CRISPR/Cas9, somatic gene transfer models) Technologien, um unser biologisches Verständnis pädiatrischer Hirntumoren zu verbessern. Ein weiterer wichtiger Fokus unserer Gruppe liegt auf der Translation der Forschungsergebnisse in die klinische Anwendung zum Wohle der Patienten. Wir sind daher federführend an zwei großen internationalen Programmen zur molekularen Diagnostik beteiligt: an der INFORM-Studie, die nach molekularen Veränderungen in den Tumoren von pädiatrischen Hochrisikopatienten sucht, die sich als Angriffspunkt für eine zielgerichtete Therapie eignen, und an der MNP2-Studie, die den Wert einer molekularen Diagnostik als Werkzeug zur akkuraten Klassifikation von Hirntumoren prüft.
Mehr Informationen über das Zentrum für pädiatrische Neuroonkologie in Heidelberg finden Sie hier.
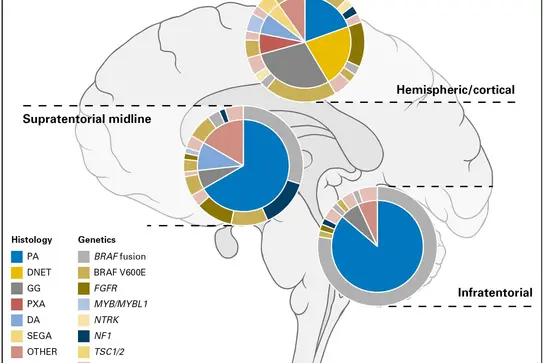
Approximation of the frequencies of different histological groupings of pediatric low-grade glioma and associated molecular genetic alterations, split by location within the brain. Adapted from Sturm D, Pfister SM and Jones DTW, J Clin Oncol 2017.
Projekte
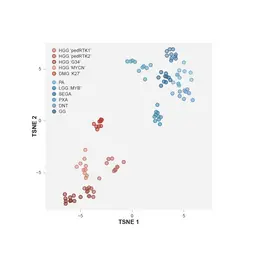
Gliome sind die häufigsten Tumore des zentralen Nervensystems (ZNS) bei Kindern und Jugendlichen. Epigenetische Analysen sind zu einem unverzichtbaren Instrument in der pädiatrischen Hirntumorforschung geworden, da sie die Diagnostik unterstützen und die molekularen Mechanismen aufdecken, die die Tumorentwicklung vorantreiben. DNA-Methylierungssignaturen, kombiniert mit histopathologischer und molekularer Tumorklassifizierung, sind weltweit anerkannt und wurden in die neuesten WHO-Leitlinien zur Klassifizierung von ZNS-Tumoren integriert. Unsere Forschung konzentriert sich auf epigenetische Modifikationen, einschließlich DNA-Methylierungsmuster und Histonveränderungen, die die Genexpression beeinflussen. Durch die Identifizierung spezifischer epigenetischer Veränderungen bei pädiatrischen Gliomen wollen wir verbesserte Diagnosewerkzeuge und zielgerichtete Therapien entwickeln. Unsere Ergebnisse haben bereits wertvolle Einblicke in die Biologie dieser Tumore geliefert und sind vielversprechend für die Entwicklung effektiverer Behandlungen in der Zukunft.
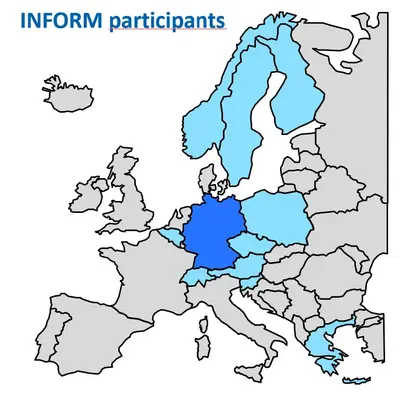
Wir spielen eine zentrale Rolle bei der Organisation des internationalen Programms für Präzisionsonkologie INFORM (INdividualized therapy FOr Relapsed Malignancies in children), indem wir molekulare Analysen und die Echtzeit-Dateninterpretation überwachen. Dies ermöglicht den Zugang zu umfassenden molekularen Profilen und einem schnellen Wirkstoff-Screening für pädiatrische Krebspatienten mit hohem Risiko in ganz Europa. Das INFORM-Netzwerk erstreckt sich über 13 europäische Länder, ist in über 100 pädiatrisch-onkologischen Forschungszentren tätig und hat mehr als 3.300 rezidivierte oder refraktäre pädiatrische Krebspatienten aufgenommen, von denen fast die Hälfte von internationalen Partnern kommt. Die erste klinische Wirkungsanalyse zeigte einen Überlebensvorteil für Patienten mit hochprioritären Zielen, die passende Behandlungen erhielten. Das Programm hat auch erfolgreich eine Plattform zur Erstellung personalisierter Ex-vivo-Wirkstoffempfindlichkeitsprofile unter realen Bedingungen integriert und liefert funktionelle Daten für die klinische Entscheidungsfindung. Es wurden erhebliche Fortschritte bei der Verbesserung der Logistik, der Methodik und der Wirksamkeit der personalisierten Diagnose und Behandlung erzielt. Dazu gehört auch der Übergang zur hochauflösenden Ganzgenomsequenzierung (WGS) als neuer Standard für die molekulare Diagnostik. Wichtig ist, dass diese Routineanalyse von den deutschen Krankenkassen erstattet wird und dazu beiträgt, ähnliche Sequenzierungs- und Screening-Plattformen in anderen Ländern einzurichten.

Niedriggradige Hirntumore sind die häufigsten Tumore des zentralen Nervensystems bei Kindern und machen etwa die Hälfte aller Hirntumore bei Kindern aus. In Deutschland und Großbritannien werden jährlich über 400 Kinder und Jugendliche diagnostiziert. Diese Tumore entstehen durch DNA-Veränderungen, die ein unkontrolliertes Zellwachstum im Gehirn oder Rückenmark verursachen. Obwohl sie langsam wachsen und sich wahrscheinlich nicht ausbreiten, ist ihre Behandlung aufgrund ihrer Lage schwierig und führt oft zu langfristigen Gesundheitsproblemen.
Das Everest Center for Low-Grade Pediatric Brain Tumors – eine Zusammenarbeit zwischen Forschungsgruppen in Berlin, Jena, London und Heidelberg (KiTZ & DKFZ) – hat sich zum Ziel gesetzt, das Verständnis zu fördern und neue Behandlungsmethoden zu entwickeln. Das 2017 gegründete Zentrum wird von The Brain Tumor Charity finanziert und hat dank der inspirierenden Spendenaktion Everest in the Alps 12 Millionen Euro (10 Millionen Pfund) erhalten.
Eine enge Partnerschaft mit dem Dana-Farber Cancer Institute in Boston stärkt die Forschungsbemühungen und stellt sicher, dass Entdeckungen junge Patienten erreichen. Darüber hinaus konzentriert sich das Projekt auf die Verbesserung der Lebensqualität, indem die Erfahrungen einzelner Patienten im Laufe der Zeit mithilfe fortschrittlicher Technologien verfolgt werden.
Der Name des Everest Center ehrt eine außergewöhnliche Spendenaktion, die von Toby, einem jungen Patienten, inspiriert wurde. Seit 2015 hat ein engagiertes Team von Skifahrern die Alpen überquert und dabei einen Höhenunterschied überwunden, der dem Mount Everest (8.848 m) entspricht, und über 10 Millionen Pfund für die Forschung gesammelt. Ihr Engagement ist von unschätzbarem Wert – im Namen aller betroffenen Familien möchten wir ihnen unseren tiefsten Dank aussprechen!

Die Dietmar Hopp Stiftung fördert den Aufbau eines internationalen klinischen „Daten-Depots“ am Hopp-Kindertumorzentrum Heidelberg (KiTZ) unter der Leitung von David Jones und Stefan Pfister. Dieses Projekt zielt darauf ab, die Grundlage dafür zu schaffen, dass Kinder und Jugendliche rasch von bereits für Erwachsene zugelassenen Krebsmedikamenten profitieren können.
Durch die Sammlung und Integration klinischer Daten aus verschiedenen internationalen Quellen soll das „Daten-Depot“ den Zugang zu wirksameren und nebenwirkungsärmeren Therapien für junge Krebspatienten beschleunigen. Dies ermöglicht es, bestehende Medikamente schneller für die Behandlung von Kindern und Jugendlichen nutzbar zu machen, ohne auf langwierige Entwicklungsprozesse neuer Medikamente warten zu müssen.
Neben dem „Daten-Depot“ wird auch die Erforschung und Entwicklung innovativer Behandlungsmethoden unterstützt, um die Heilungschancen und Lebensqualität junger Patienten zu verbessern.

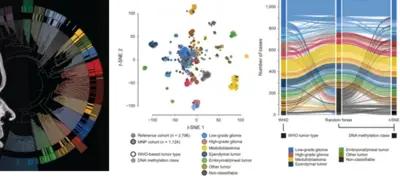
MNP Int-R ist eine Fortführung und Folgestudie von MNP 2.0, in der die Verwendung von DNA-Methylierungssignaturen als Teil einer integrierten histologischen und molekularen Klassifizierung zur Verbesserung der Genauigkeit von pädiatrischen Hirntumor-Diagnosen bei der Erstdiagnose etabliert wurde. Diese Initiative ebnete den Weg für die Einbeziehung der molekularen Diagnostik, einschließlich der DNA-Methylierungsprofilierung und der gezielten Genpanel-Sequenzierung, in die Routinediagnostik für primäre ZNS-Tumoren bei Kindern und Jugendlichen. Die integrierte Referenzanalyse hilft bei der Identifizierung bisher unbekannter Tumorarten und -subtypen, der Aufdeckung ihrer biologischen Zusammenhänge und der Klassifizierung seltener molekular definierter Tumorentitäten. Die weitere Verfeinerung und Schulung des auf DNA-Methylierung basierenden Klassifizierungssystems verbessert die molekulare Diagnostik und prägt die Gestaltung zukünftiger therapeutischer Studien, wodurch personalisierte Präzisionsbehandlungen und neuartige zielgerichtete Therapieansätze vorangetrieben werden. Durch die Integration von pathologischen, radiologischen und molekularen Daten in eine umfassende Tumordiagnose zielt MNP Int-R darauf ab, hohe diagnostische Standards in einem internationalen Umfeld aufrechtzuerhalten und das klinische Krankheitsmanagement erheblich zu beeinflussen. Darüber hinaus wird die fortlaufende Datenerhebung die diagnostische Genauigkeit und die Behandlungsstrategien weiter verfeinern.
Unser Team
Gruppenbild
Publikationen
2019 - 2025
Mehr erfahrenErnst, K.
Establishment and evaluation of single-cell RNA-sequencing methods to investigate tumor heterogeneity in newly generated pediatric glioma models
Deng, M. Y.
Molecular identification and characterization of novel pediatric brain tumor entities through genome-wide DNA methylation profiling
Sommerkamp, A.
Molecular comparison, preclinical modeling and improved diagnostics of pediatric low-grade gliomas
Ismer, B.
Novel gene fusions identified as new drug targets in paediatric glioma and their pre-clinical characterization
Maile, J.
Profiling of epigenetic-related vulnerabilities in K27M diffuse midline glioma using CRISPR/Cas9 genetic screening
Ausgewählte Publikationen
Benfatto S, Sill M, Jones DTW, Pfister SM, Sahm F, von Deimling A, Capper D, Hovestadt V
van Tilburg CM, Kilburn LB, Perreault S, Schmidt R, Azizi AA, Cruz-Martínez O, Zápotocký M, Scheinemann K, Meeteren AYNS, Sehested A, Opocher E, Driever PH, Avula S, Ziegler DS, Capper D, Koch A, Sahm F, Qiu J, Tsao LP, Blackman SC, Manley P, Milde T, Witt R, Jones DTW, Hargrave D, Witt O.
Sturm D, Andreiuolo F, Gessi M, Kölsche C, …, Pietsch T*, Sahm F*, Pfister SM*, Jones DTW*
FOXR2 is an epigenetically regulated pan-cancer oncogene that activates ETS transcriptional circuits
Tsai JW, Cejas P, Wang DK, Patel S, …, Long H, Jones DTW*, Bandopadhayay P*, Phoenix TN*
Deng MY, Sturm D, Pfaff E, Sill M, …, Korshunov A, von Deimling A, Pfister SM, Jones DTW
Clarke M, Mackay A, Ismer B, Pickles JC, …, Ellison DW*, Jacques TS*, Jones DTW*, Jones C*
Jones DTW, Banito A, Grünewald TGP, …, Schleiermacher G, Smith MA, Westermann F, Pfister SM
Capper D*, Jones DTW*, Sill M*, Hovestadt V*, …, Brandner S, Korshunov A, von Deimling A, Pfister SM
Gröbner SN, Worst BC, Weischenfeldt J, Buchhalter I, …, Jones DTW, Lichter P, Chavez L, Zapatka M, Pfister SM
Mackay A, Burford A, Carvalho D, Izquierdo E, …, Jones DTW, Fouladi M, von Bueren AO, Baudis M, Resnick A, Jones C
Bender S, Gronych J, Warnatz H-J, Hutter B, …, Yaspo ML, Pfister SM*, Lichter P*, Jones DTW*
Worst BC, van Tilburg CM, Balasubramanian GP, Fiesel P, …, Capper D, Pfister SM*, Jones DTW* and Witt O*
Bender S, Tang Y, Lindroth AM, Hovestadt V, Jones DTW, …., Monje M, Plass C, Cho YJ, Pfister SM
Alexandrov LB, Nik-Zainal S, Wedge DC, Aparicio SA, …, Shibata T, Pfister SM, Campbell PJ, Stratton MR
Jones DTW*, Hutter B*, Jäger N*, Korshunov A, …, Jabado N, Eils R, Lichter P and Pfister SM
Schwartzentruber J*, Korshunov A*, Liu XY*, Jones DTW, …, Plass C, Majewski J, Pfister SM, Jabado N
Jones DT, Kocialkowski S, Liu L, Pearson DM, Bäcklund LM, Ichimura K, Collins VP
Kontaktieren Sie uns



