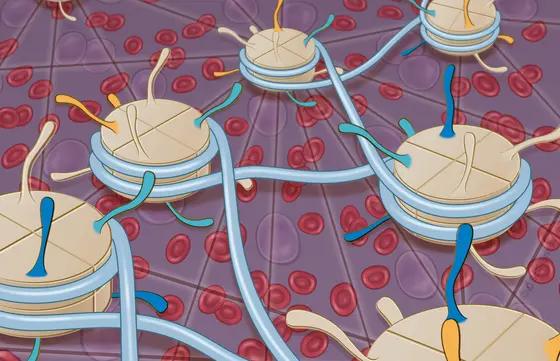
In Krebszellen ist nicht nur die DNA durch Mutationen verändert, sondern auch die am Genom angehefteten epigenetischen Signale. Darunter versteht man an die DNA oder die DNA-Bindeproteine (Histone) reversibel gekoppelte kleine chemische Gruppen (z.B. durch Methyl- oder Azetylgruppen). Falsche epigenetische Signale können bewirken, dass die Erbinformation nicht richtig gelesen oder in ihrer Funktion unterdrückt wird. Dies kann dazu führen, dass eine kranke Zelle einen Selbstzerstörungsbefehl ignoriert oder Gene ausschaltet, die die Krebsentstehung verhindern.
„In unserer aktuellen Studie haben wir eine fast vollständige Karte der wichtigsten epigenetischen Signale erstellt, die bei der chronischen lymphatischen Leukämie (CLL) gegenüber gesunden Zellen verändert sind“, erläutert Karsten Rippe, der zusammen mit Daniel Mertens vom DKFZ den Forschungsverbund zur Untersuchung der CLL Epigenetik koordiniert.
„Die Stärke unserer Arbeit liegt in der Verknüpfung biomedizinischer Grundlagenforschung, die am DKFZ ihren Schwerpunkt hat, und klinischer Forschung“, so Daniel Mertens, der gleichzeitig auch am Ulmer Klinikum arbeitet. „Dabei haben wir experimentelle Arbeiten im Labor mit Analysen und Modellierungen am Computer integriert und konnten so mathematische Modelle entwickeln, die vorhersagen, wie sich die Genaktivität in CLL-Zellen verändert.“ Sind beispielsweise in den Krebszellen eines Patienten relevante „Krebsbremsen“ (Tumor-Suppressorgene) in ihrer Aktivität unterdrückt, so hat dies erheblichen Einfluss auf die Erkrankung.
Als nächstes wollen die Wissenschaftler herausfinden, wie Medikamente das epigenetische Programm der Zelle beeinflussen und wie man mit Hilfe der Untersuchung epigenetischer Signale den Behandlungserfolg vorhersagen kann.
Die Arbeit wurde vom Bundesministerium für Bildung und Forschung gefördert.
Mallm JP, Iskar M, Ishaque N, Klett LC, Kugler SJ, Muino JM, Teif VB, Poos AM, Großmann S, Erdel F, Tavernari D, Koser SD, Schumacher S, Brors B, König R, Remondini D, Vingron M, Stilgenbauer S, Lichter P, Zapatka M, Mertens D & Rippe K (2019) Linking aberrant chromatin features in chronic lymphocytic leukemia to deregulated transcription factor networks. Mol Syst Biol 5, e8339. doi: 10.15252/msb.20188339.
Ein Bild zur Meldung steht zur Verfügung unter: MSB_cover_Vol15.jpg
BU: Ein Netzwerk von falschen epigenetischen Signalen an den Histonproteinen und der DNA führt in Zellen der chronisch-lymphatischen Leukämie zu Fehlern beim Auslesen der Erbinformation.
Nutzungshinweis für Bildmaterial zu Pressemitteilungen
Die Nutzung ist kostenlos. Das Deutsche Krebsforschungszentrum (DKFZ) gestattet die einmalige Verwendung in Zusammenhang mit der Berichterstattung über das Thema der Pressemitteilung bzw. über das DKFZ allgemein. Bitte geben Sie als Bildnachweis an: „Quelle: Molecular Systems Biology, Illustration von SciStories“.
Eine Weitergabe des Bildmaterials an Dritte ist nur nach vorheriger Rücksprache mit der DKFZ-Pressestelle (Tel. 06221 42 2854, E-Mail: presse@dkfz.de) gestattet. Eine Nutzung zu kommerziellen Zwecken ist untersagt.