Fortgeschrittene Tumoren sind durch eine große Vielfalt an Erbgutdefekten gekennzeichnet. Während bei vielen Krebsarten tausende kleiner „Schreibfehler“ im Erbgut zu veränderten, funktionsunfähigen Proteinen führen, sind für das Prostatakarzinom größere Erbgutdefekte charakteristisch. Ganze DNA-Abschnitte sind verloren, verdoppelt oder verkehrt herum angeordnet. Zusätzlich zu diesen so genannten strukturellen Defekten zeichnet sich Prostatakrebs durch große Unterschiede im Muster der DNA-Methylierung aus. Mit diesen kleinen chemischen Anhängseln, die zu den epigenetischen Mechanismen zählen, reguliert die Zelle unter anderem, welche ihrer Gene abgelesen werden.
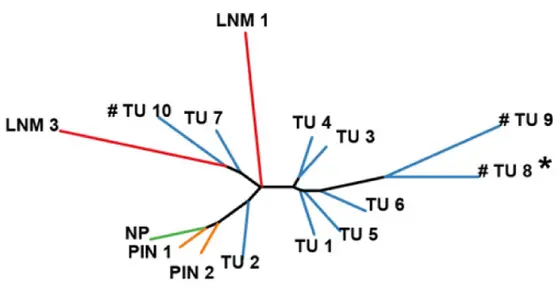
Die strukturellen Erbgutveränderungen, die sich im Laufe der Krebsentstehung ansammeln, führen zu einer „Evolution“ der Krebszellen, so dass sich ein fortgeschrittener Tumor aus einer Gruppe von verschiedenen „Tochterklonen“ zusammensetzt. Das bedeutet, dass nicht nur jeder Prostatatumor einzigartig ist, sondern auch, dass derselbe Tumor aus verschiedenen Klonen besteht, die sich in klinischen Aspekten unterscheiden können, etwa in der Therapieresistenz.
Wissenschaftler um Christoph Plass im Deutschen Krebsforschungszentrum und Mitglieder des deutschen ICGC Konsortiums “Early onset prostate cancer“ untersuchten nun, ob sich auch anhand der epigenetischen Veränderungen die Evolution des Tumors und somit auch die Zusammensetzung der verschiedenen Tochterklone nachvollziehen lässt. Dazu analysierten sie bei fünf Fällen von Prostatakrebs simultan sowohl die Gene als auch deren Methylierung.
Die Forscher verglichen Gewebeproben von verschiedenen Stellen eines Tumors mit noch nicht völlig entartetem Gewebe aus der Tumorumgebung und mit Lymphknotenmetastasen. Sie zeigten, dass sowohl die strukturellen Genomveränderungen als auch die Änderungen im Methylierungsmuster gleichermaßen die Evolution der einzelnen Tumor-Klone abbildeten. Offenbar verläuft die Evolution des Epigenoms parallel mit dem Auftauchen neuer struktureller Erbgutveränderungen.
Wichtig ist die Beobachtung, dass Metastasen nicht zwangsläufig am „Ende“ der Entwicklung eines Tumors entstehen: So fehlen in einem Fall den Metastasen die Chromosomendefekte, die alle anderen Gewebeproben dieses Tumors kennzeichnen, was auf eine frühere Entstehung der Tochtergeschwülste schließen lässt. Bei einigen der untersuchten Fälle entwickelten sich die Metastasen aus einem gemeinsamen Vorfahren, bei anderen hingegen aus verschiedenen Tochterklonen. Allgemein gilt, dass Metastasen immer Merkmale tragen, die den anderen Tumorklonen fehlen. In der Regel betreffen diese epigenetischen oder genetischen Veränderungen Gene, die metastasierenden Krebszellen ihre typischen Eigenschaften verleihen.
Die epigenetischen Unterschiede zwischen den Tochterklonen sind nicht gleichmäßig über das gesamte Tumorerbgut verteilt. In den meisten Fällen betreffen sie besonders solche Bereiche, die für prostataspezifische Prozesse wichtig sind. Dazu zählen etwa diejenigen „Gen-Verstärker“ (Enhancer), die von den Rezeptoren für das männliche Geschlechtshormon Androgen gesteuert werden. Da sich die verschiedenen Methylierungsmuster auf die Genaktivität auswirken, ist es naheliegend, dass sich die Tochterklone in der Art und Weise, wie sie Androgensignale verarbeiten, stark unterscheiden.
„Die genetische und die epigenetische Evolution der Prostatatumoren verläuft unabhängig voneinander, kommt aber zu demselben Ziel“, erklärt Dr. Clarissa Gerhäuser. „Deshalb kann der technisch weniger aufwändige Nachweis der epigenetischen Evolution einzelner Tumorklone dabei helfen, schnell und genau funktionelle Informationen zu erhalten, die für klinische Entscheidungen relevant sein können.“ Die Wissenschaftler gehen davon aus, dass durch genaueres Verständnis der genetischen und epigenetischen Vielfalt innerhalb eines Tumors bessere Behandlungserfolge erzielt werden können.
David Brocks, Yassen Assenov, Sarah Minner, Olga Bogatyrova, Ronald Simon, Christina Koop, Christopher Oakes, Manuela Zucknick, Daniel Bernhard Lipka, Joachim Weischenfeldt, Lars Feuerbach, Richard Cowper-Sallari, Mathieu Lupien, Benedikt Brors, Jan Korbel, Thorsten Schlomm, Amos Tanay, Guido Sauter, Clarissa Gerhäuser und Christoph Plass: Epigenetic Intratumor Heterogeneity reflects Clonal Evolution in Aggressive Prostate Cancer. Cell Reports 2014, DOI: 10.1016/j.celrep.2014.06.053
Über das DKFZ
Das Deutsche Krebsforschungszentrum (DKFZ) ist mit mehr als 3.000 Mitarbeiterinnen und Mitarbeitern die größte biomedizinische Forschungseinrichtung in Deutschland. Wissenschaftlerinnen und Wissenschaftler erforschen im DKFZ, wie Krebs entsteht, erfassen Krebsrisikofaktoren und suchen nach neuen Strategien, die verhindern, dass Menschen an Krebs erkranken. Sie entwickeln neue Methoden, mit denen Tumoren präziser diagnostiziert und Krebspatienten erfolgreicher behandelt werden können. Beim Krebsinformationsdienst (KID) des DKFZ erhalten Betroffene, Interessierte und Fachkreise individuelle Antworten auf alle Fragen zum Thema Krebs.
Um vielversprechende Ansätze aus der Krebsforschung in die Klinik zu übertragen und so die Chancen von Patientinnen und Patienten zu verbessern, betreibt das DKFZ gemeinsam mit exzellenten Universitätskliniken und Forschungseinrichtungen in ganz Deutschland Translationszentren:
- Nationales Centrum für Tumorerkrankungen (NCT, 6 Standorte)
- Deutsches Konsortium für Translationale Krebsforschung (DKTK, 8 Standorte)
- Hopp-Kindertumorzentrum (KiTZ) Heidelberg
- Helmholtz-Institut für translationale Onkologie (HI-TRON) Mainz – ein Helmholtz-Institut des DKFZ
- DKFZ-Hector Krebsinstitut an der Universitätsmedizin Mannheim
- Nationales Krebspräventionszentrum (gemeinsam mit der Deutschen Krebshilfe)
Das DKFZ wird zu 90 Prozent vom Bundesministerium für Forschung, Technologie und Raumfahrt und zu 10 Prozent vom Land Baden-Württemberg finanziert und ist Mitglied in der Helmholtz-Gemeinschaft Deutscher Forschungszentren.