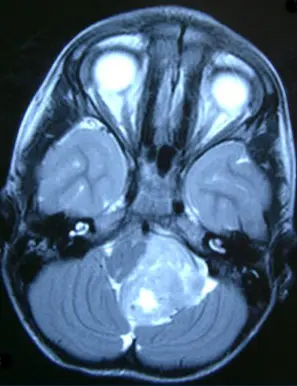
Ependymome sind eine Gruppe von Hirntumoren, die bei Kindern und Erwachsenen gleichermaßen auftreten. Sie entstehen in allen Bereichen des zentralen Nervensystems, im Rückenmark, im Kleinhirn und im Großhirn. Neben ihren verschiedenen Lokalisationen unterscheiden sich Ependymome auch in ihrer Biologie und ihrem klinischen Verlauf.
Um die Tumoren einzuteilen, steht Ärzten derzeit nur die histopathologische Untersuchung zur Verfügung, die jedoch unzuverlässige Aussagen über den Verlauf und Biologie der Erkrankung liefert. Um eine aussagekräftigere Klassifikation der Tumoren zu erreichen, haben Kristian Pajtler und Kollegen um Dr. Marcel Kool und Prof. Dr. Stefan Pfister im Deutschen Krebsforschungszentrum nun 500 Ependymome sorgfältig molekular charakterisiert. Dabei untersuchten sie neben epigenetischen Merkmalen wie der DNA-Methylierung auch genetische Abweichungen und analysierten die Genaktivität.
Die untersuchten Tumoren umfassten alle histologischen Einteilungen und stammen von Patienten aller Altersgruppen aus allen Gehirnregionen, so dass eine umfassende Klassifizierung dieser heterogenen Gruppe von Krebserkrankungen möglich war.
Bei der Auswertung der Methylierungsmuster entdeckten die Forscher, dass sich die Tumoren in neun verschiedene Gruppen einteilen lassen. Jedem der drei ZNS-Bereiche, in denen Ependymome auftreten (Rückenmark, Kleinhirn und Großhirn) ließen sich drei dieser Gruppen zuordnen.
Zusätzlich zu den jeweiligen Methylierungsprofilen unterscheiden sich die neun Gruppen in ihrer Genetik, ihrer Genaktivität, im Erkrankungsalter und im klinischen Verlauf. „Die Erkrankungen sind so unterschiedlich, dass wir fast davon ausgehen müssen, dass sie aus verschiedenen Zelltypen entstehen“, spekuliert der Kinderonkologe und Molekularbiologe Stefan Pfister.
Die Methylierungsmuster bleiben im Verlauf der Erkrankung stabil, auch bei einem Rückfall, was die Klassifizierung der Erkrankung zusätzlich unterstützt. Da für die molekulare Analyse winzigste DNA-Mengen ausreichen, die sogar von paraffineingebetteten Gewebeproben stammen können, eignet sich der Nachweis gut für die klinische Routine.
Die molekulare Analyse bildet die verschiedenen Verläufe der Erkrankung präziser ab als die Einteilung anhand der Gewebebeurteilung am Mikroskop. „Dass die Patienten mit besonders ungünstiger Prognose, zumeist Kinder, hauptsächlich zwei der neun Gruppen zugeordnet werden können, ist ein sehr gutes Indiz dafür, dass unsere molekulare Einteilung klinisch relevant ist“, sagt Studienleiter Marcel Kool.
Bei den Analysen der Genaktivität entdeckten die Forscher auch, dass in den Tumorzellen der verschiedenen Gruppen unterschiedliche Signalwege überaktiv sind. Einig der Schlüsselproteine dieser Signalwege lassen sich mit zielgerichteten Wirkstoffen blockieren, was das Tumorwachstum bremst. „Gerade in den beiden Gruppen der Tumoren, für die wir heute noch keine zufriedenstellende Behandlungsmöglichkeit haben, könnte die Analyse der DNA-Methylierung der Schlüssel für neue Therapien sein,“ sagt Stefan Pfister.
Kristian W. Pajtler, Hendrik Witt, Martin Sill, David T.W. Jones, Volker Hovestadt, Fabian Kratochwil, Khalida Wani, Ruth Tatevossian, Chandanamali Punchihewa, Pascal Johann, Jüri Reimand, Hans-Jörg Warnatz, Marina Ryzhova, Steve Mack, Vijay Ramaswamy, David Capper, Leonille Schweizer, Laura Sieber, Andrea Wittmann, Zhiqin Huang, Peter van Sluis, Richard Volckmann, Jan Koster, Rogier Versteeg, Daniel Fults, Helen Toledano, Smadar Avigad, Lindsey M. Hoffman, Andrew M. Donson, Nicholas Foreman, Ekkehard Hewer, Karel Zitterbart, Mark Gilbert, Terri S. Armstrong, Nalin Gupta, Jeffrey C. Allen, Matthias A. Karajannis, David Zagzag, Martin Hasselblatt, Andreas E. Kulozik, Olaf Witt. Peter Collins, Katja von Hoff, Stefan Rutkowski, Torsten Pietsch, Gary Bader, Marie-Laure Yaspo, Andreas von Deimling, Peter Lichter, Michael D. Taylor, Richard Gilbertson, David W. Ellison, Kenneth Aldape, Andrey Korshunov, Marcel Kool und Stefan M. Pfister:
Molecular Classification of Ependymal Tumors across all CNS Compartments, Histopathological Grades and Age Groups. Cancer Cell 27, 1-16, 2015, DOI: 10.1016/j.ccell.2015.04.002