Spatially Resolved Omics
Heterogeneity of tumor cells within their endogenous "microenvironment" of different types of non-malignant cells is emerging as an important aspect of personalized oncology. This information, however, is lost in the single cell sequencing analysis of isolated cell suspension or nuclei. Recent technological advancements make it now possible to combine different "omics" methods with microscopy imaging. This allows single-cell sequencing, proteomics or metabolomics analysis to be integrated with microscopy phenotypes through sequencing or mass spectrometry, opening up entirely new opportunities in cancer research. Via joint activities with the DKFZ Single Cell Open Lab (link) and the Light Microscopy Core Facility (link) as well as the CellNetworks Spatial Transcriptomics Lab and the Explorer START-HD program of the Heidelberg Molecular Life Science (HMLS) program (https://www.uni-heidelberg.de/excellenceinitiative/institutionalstrategy/hmls.html) these technologies are developed and applied. They currently involve multiplex single molecule RNA-FISH (smRNA-FISH), "Slide-seq" (capturing RNA after imaging and lysis of cells on slides), multiplex immunostaining, mass spectrometry imaging and spatially resolved metabolomics (SpaceM).
Multiplex Single Molecule FISH

© dkfz.de
Combinatorial Color Barcoding For Multiplexing Single Molecule RNA Visualization. For further details see Cheng et al 2017 bioRxiv 127373.
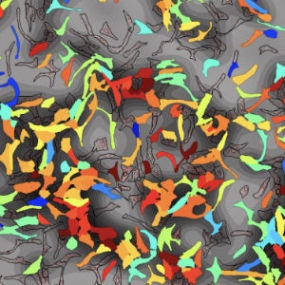
Spatially resolved metabolomics (SpaceM)
© dkfz.de
Spatial single-cell profiling of intracellular metabolomes in situ. For further details see Rappez et al 2019, bioRxiv 510222.
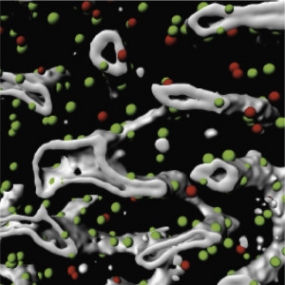
Spatially resolved transcriptomics of the bone marrow
© dkfz.de
Combined single-cell and spatial transcriptomics reveals the molecular, cellular and spatial bone marrow niche organization. For further details see Baccin et al 2019 bioRxiv 718395.
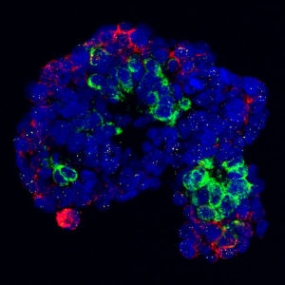
Transcriptome analysis of 3D cell models
© dkfz.de
Visual features and gene expression in 3D cell culture systems are linked. For further details see Tirier et al 2019 Sci Rep 9, 12367
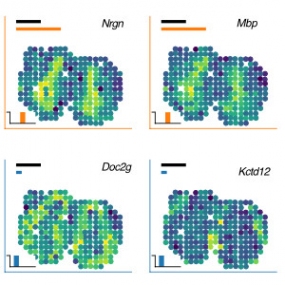
Identification of spatially variable genes
© dkfz.de
The SpatialDE methods provides a statistical test to identify genes with spatial patterns of expression variation from multiplexed imaging or spatial RNA-sequencing data. For further details see Svensson et al 2018 Nat Meth 15, 343
Contact
Mathias Heikenwälder (F180)
Hakun Liu (A240)
Karsten Rippe, Division of Chromatin Networks (B066)
